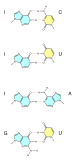
Ζεύγος βάσεων

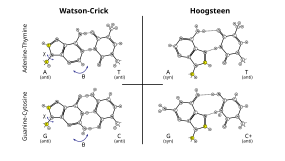
Ένα ζεύγος βάσεων (base pair, bp) είναι μια θεμελιώδης μονάδα δίκλωνων νουκλεϊκών οξέων που αποτελείται από δύο νουκλεοβάσεις που συνδέονται μεταξύ τους με δεσμούς υδρογόνου. Αποτελούν τα δομικά στοιχεία της διπλής έλικας του DNA και συμβάλλουν στη διπλωμένη δομή τόσο του DNA όσο και του RNA. Υπαγορεύεται από συγκεκριμένα μοτίβα δεσμών υδρογόνου, τα ζεύγη βάσεων Watson–Crick (ή Watson–Crick–Franklin) (γουανίνη–κυτοσίνη (GC) και αδενίνη–θυμίνη)) (AT)[1] που επιτρέπουν στην έλικα του DNA να διατηρεί μια κανονική ελικοειδή δομή που εξαρτάται επιδέξια από τη νουκλεοτιδική αλληλουχία τους.[2] Η συμπληρωματική φύση αυτής της δομής με βάση τα ζεύγη παρέχει ένα εφεδρικό αντίγραφο της γενετικής πληροφορίας που κωδικοποιείται σε κάθε κλώνο DNA. Η κανονική δομή και η εφεδρικότητα των δεδομένων που παρέχονται από τη διπλή έλικα του DNA καθιστούν το DNA κατάλληλο για την αποθήκευση γενετικών πληροφοριών, ενώ το ζευγάρωμα βάσεων μεταξύ DNA και εισερχόμενων νουκλεοτιδίων παρέχει τον μηχανισμό μέσω του οποίου η DNA πολυμεράση αντιγράφει DNA και η RNA πολυμεράση μεταγράφει το DNA σε RNA. Πολλές πρωτεΐνες που δεσμεύουν το DNA μπορούν να αναγνωρίσουν συγκεκριμένα μοτίβα ζευγαρώματος βάσεων που προσδιορίζουν συγκεκριμένες ρυθμιστικές περιοχές γονιδίων. Ενδομοριακά ζεύγη βάσεων μπορούν να εμφανιστούν εντός μονόκλωνων νουκλεϊκών οξέων. Αυτό είναι ιδιαίτερα σημαντικό στα μόρια RNA (π.χ., μεταφορικό RNA), όπου τα ζεύγη βάσεων Watson-Crick (γουανίνη-κυτοσίνη και αδενίνη-ουρακίλη) επιτρέπουν το σχηματισμό κοντών δίκλωνων ελίκων, και μια ευρεία ποικιλία αλληλεπιδράσεων που δεν είναι Watson-Crick (π.χ. G-U ή A-A) που επιτρέπουν στα RNA να αναδιπλωθούν σε τεράστιο εύρος συγκεκριμένων τρισδιάστατων δομών RNA. Επιπλέον, το ζευγάρωμα βάσεων μεταξύ μεταφορικού RNA (tRNA) και αγγελιοφόρου RNA (mRNA) αποτελεί τη βάση για τα συμβάντα μοριακής αναγνώρισης που έχουν ως αποτέλεσμα η αλληλουχία νουκλεοτιδίων του mRNA να μεταφραστεί στην αλληλουχία αμινοξέων της πρωτεΐνης μέσω του γενετικού κώδικα. Το μέγεθος ενός μεμονωμένου γονιδίου ή ολόκληρου του γονιδιώματος ενός οργανισμού μετριέται συχνά σε ζεύγη βάσεων, επειδή το DNA είναι συνήθως δίκλωνο. Ως εκ τούτου, ο αριθμός των συνολικών ζευγών βάσεων είναι ίσος με τον αριθμό των νουκλεοτιδίων σε έναν από τους κλώνους (με εξαίρεση τις μη κωδικοποιητικές μονόκλωνες περιοχές των τελομερών). Το απλοειδές ανθρώπινο γονιδίωμα (23 χρωμοσώματα) εκτιμάται ότι έχει μήκος περίπου 3,2 δισεκατομμύρια ζεύγη βάσεων και περιέχει 20.000–25.000 διακριτά γονίδια που κωδικοποιούν πρωτεΐνες.[3][4][5][6] Μια kilobase (kb) είναι μια μονάδα μέτρησης στη μοριακή βιολογία ίση με 1000 ζεύγη βάσεων DNA ή RNA.[7] Ο συνολικός αριθμός ζευγών βάσεων DNA στη Γη υπολογίζεται σε 5,0 × 1037 με βάρος 50 δισεκατομμύρια τόνους.[8]Συγκριτικά, η συνολική μάζα της βιόσφαιρας έχει υπολογιστεί ότι είναι έως και 4 τρισεκατομμύρια τόνοι άνθρακα).[9]
Δεσμοί υδρογόνου και σταθερότητα
[Επεξεργασία | επεξεργασία κώδικα]
|

|
Ο δεσμός υδρογόνου είναι η χημική αλληλεπίδραση που διέπει τους κανόνες του ζεύγους βάσεων που περιγράφονται παραπάνω. Η κατάλληλη γεωμετρική αντιστοιχία δοτών και αποδεκτών δεσμών υδρογόνου επιτρέπει να σχηματίζονται σταθερά μόνο τα σωστά ζεύγη. Το DNA με υψηλή περιεκτικότητα σε GC (γουανίνη-κυτοσίνη) είναι πιο σταθερό από το DNA με χαμηλή περιεκτικότητα σε GC. Κυρίως, ωστόσο, οι αλληλεπιδράσεις στοίβαξης είναι κυρίως υπεύθυνες για τη σταθεροποίηση της δομής διπλής ελίκωσης. Η συμβολή του ζεύγους βάσεων Watson-Crick στην παγκόσμια δομική σταθερότητα είναι ελάχιστη, αλλά ο ρόλος του στην εξειδίκευση που κρύβεται πίσω από τη συμπληρωματικότητα είναι, αντίθετα, μέγιστης σημασίας, καθώς αυτό βασίζεται στις εξαρτώμενες από το πρότυπο διαδικασίες του κεντρικού δόγματος (π.χ. αντιγραφή του DNA).[10] Οι μεγαλύτερες νουκλεοβάσεις, η αδενίνη και η γουανίνη, είναι μέλη μιας κατηγορίας χημικών δομών διπλού δακτυλίου που ονομάζονται πουρίνες. Οι μικρότερες νουκλεοβάσεις, η κυτοσίνη και η θυμίνη (και η ουρακίλη), είναι μέλη μιας κατηγορίας χημικών δομών ενός δακτυλίου που ονομάζονται πυριμιδίνες. Οι πουρίνες είναι συμπληρωματικές μόνο με τις πυριμιδίνες: τα ζεύγη πυριμιδίνης-πυριμιδίνης είναι ενεργειακά δυσμενή επειδή τα μόρια είναι πολύ μακριά μεταξύ τους για να δημιουργηθεί δεσμός υδρογόνου. Τα ζεύγη πουρίνης-πουρίνης είναι ενεργειακά δυσμενή επειδή τα μόρια είναι πολύ κοντά, οδηγώντας σε επικάλυψη απώθησης. Το ζεύγος βάσεων πουρίνης-πυριμιδίνης της AT (αδενίνης-θυμίνης) ή της GC (γουανίνης-κυτοσίνης) ή της UA (ουρακίλης-αδενίνης) (στο RNA) έχει ως αποτέλεσμα τη σωστή διπλή δομή. Τα μόνα άλλα ζεύγη πουρίνης-πυριμιδίνης θα ήταν τα AC και GT και UG (στο RNA). Αυτά τα ζεύγη είναι αναντιστοιχα επειδή τα πρότυπα των δοτών και των αποδεκτών υδρογόνου δεν αντιστοιχούν. Η σύζευξη GU, με δύο δεσμούς υδρογόνου, συμβαίνει αρκετά συχνά στο RNA. Τα ζευγαρωμένα μόρια DNA και RNA είναι συγκριτικά σταθερά σε θερμοκρασία δωματίου, αλλά οι δύο κλώνοι νουκλεοτιδίων θα διαχωριστούν πάνω από ένα σημείο τήξης που καθορίζεται από το μήκος των μορίων, την έκταση της εσφαλμένης σύζευξης (εάν υπάρχει) και το περιεχόμενο GC. Υψηλότερη περιεκτικότητα σε GC οδηγεί σε υψηλότερες θερμοκρασίες τήξης. Επομένως, δεν προκαλεί έκπληξη το γεγονός ότι τα γονιδιώματα ακραιοφίλων οργανισμών όπως ο Thermus thermophilus είναι ιδιαίτερα πλούσια σε GC. Αντίθετα, οι περιοχές ενός γονιδιώματος που πρέπει να διαχωρίζονται συχνά — για παράδειγμα, οι περιοχές προαγωγέα για συχνά — μεταγραφόμενα γονίδια — είναι συγκριτικά φτωχές σε GC. Η περιεκτικότητα σε GC και η θερμοκρασία τήξης πρέπει επίσης να λαμβάνονται υπόψη κατά τον σχεδιασμό εκκινητών αντιδράσεων PCR.
Παραδείγματα
[Επεξεργασία | επεξεργασία κώδικα]Οι ακόλουθες αλληλουχίες DNA απεικονίζουν μοτίβα δίκλωνου ζεύγους. Κατά σύμβαση, ο επάνω κλώνος γράφεται από το 5'-άκρο προς το 3'-άκρο. Έτσι, ο κάτω κλώνος γράφεται 3′ προς το 5′.
- Μια αλληλουχία DNA με ζεύγη βάσεων:
ATCGATTGAGCTCTAGCGTAGCTAACTCGAGATCGC
- Η αντίστοιχη αλληλουχία RNA, στην οποία η ουρακίλη αντικαθιστά τη θυμίνη στον κλώνο RNA:
AUCGAUUGAGCUCUAGCGUAGCUAACUCGAGAUCGC
Ανάλογα βάσης και παρεμβολείς
[Επεξεργασία | επεξεργασία κώδικα]Τα χημικά ανάλογα των νουκλεοτιδίων μπορούν να αντικαταστήσουν τα κατάλληλα νουκλεοτίδια και να δημιουργήσουν μη κανονικό ζεύγος βάσεων, οδηγώντας σε σφάλματα (κυρίως σημειακές μεταλλάξεις) στην αντιγραφή του DNA και την Μεταγραφή του DNA . Αυτό οφείλεται στην ισοστερική χημεία τους. Ένα κοινό μεταλλαξιογόνο ανάλογο βάσης είναι η 5-βρωμοουρακίλη, η οποία μοιάζει με τη θυμίνη, αλλά μπορεί να ζευγαρώσει με τη γουανίνη στη μορφή της ενόλης.[11] Άλλες χημικές ουσίες, γνωστές ως παρεμβολείς DNA (DNA intercalators), ταιριάζουν στο κενό μεταξύ γειτονικών βάσεων σε ένα μόνο κλώνο και προκαλούν μεταλλάξεις μετατόπισης πλαισίου (frameshift mutations) μεταμφιεσμένοι ως βάση, προκαλώντας την παράλειψη του μηχανισμού αντιγραφής του DNA ή την εισαγωγή επιπλέον νουκλεοτιδίων στην παρεμβαλλόμενη θέση. Οι περισσότεροι παρεμβολείς είναι μεγάλες πολυαρωματικές ενώσεις και είναι γνωστές ή ύποπτες καρκινογόνες.Τα παραδείγματα περιλαμβάνουν το βρωμιούχο αιθίδιο και την ακριδίνη.[12]
Επιδιόρθωση αταίριαστου ζεύγους
[Επεξεργασία | επεξεργασία κώδικα]Τα αταίριαστα ζεύγη βάσεων μπορούν να δημιουργηθούν από σφάλματα της αντιγραφή του DNA και ως ενδιάμεσα κατά τη διάρκεια του ομόλογου ανασυνδυασμού. Η διαδικασία επιδιόρθωσης ασυμφωνίας πρέπει κανονικά να αναγνωρίζει και να επιδιορθώνει σωστά έναν μικρό αριθμό αστοχιών βάσεων μέσα σε μια μακρά ακολουθία κανονικών ζευγών βάσεων DNA. Για να επιδιορθωθούν οι αναντιστοιχίες που σχηματίστηκαν κατά την αντιγραφή του DNA, έχουν εξελιχθεί αρκετές διακριτές διεργασίες επιδιόρθωσης για τη διάκριση μεταξύ του κλώνου μήτρας και του νεοσχηματισμένου κλώνου, έτσι ώστε να αφαιρείται μόνο το πρόσφατα εισαχθέν εσφαλμένο νουκλεοτίδιο (προκειμένου να αποφευχθεί η δημιουργία μετάλλαξης).[13]
Μετρήσεις μήκους
[Επεξεργασία | επεξεργασία κώδικα]
:
Οι ακόλουθες συντομογραφίες χρησιμοποιούνται συνήθως για να περιγράψουν το μήκος ενός μορίου DNA|RNA:
- bp = ζεύγος βάσεων—ένα bp αντιστοιχεί περίπου σε 3,4 Å (340 pm)[14] μήκους κατά μήκος του κλώνου και σε περίπου 618 ή 643 daltons για το DNA και το RNA αντίστοιχα.
- kb (= kbp) = kilo–ζεύγος βάσεων = 1,000 bp
- Mb (= Mbp) = mega– ζεύγος βάσεων = 1,000,000 bp
- Gb (= Gbp) = giga– ζεύγος βάσεων = 1,000,000,000 bp
Για μονόκλωνο DNA/RNA, χρησιμοποιούνται μονάδες νουκλεοτιδίων—συντομογραφία nt (ή knt, Mnt, Gnt αντίστοιχα)—καθώς δεν είναι ζευγαρωμένα. Για τη διάκριση μεταξύ μονάδων μνήμης υπολογιστή και βάσεων, μπορούν να χρησιμοποιηθούν kbp, Mbp, Gbp, κ.λπ. για ζεύγη βάσεων. Το centimorgan χρησιμοποιείται επίσης συχνά για να υποδηλώσει απόσταση κατά μήκος ενός χρωμοσώματος, αλλά ο αριθμός των ζευγών βάσεων στα οποία αντιστοιχεί ποικίλλει ευρέως. Στο ανθρώπινο γονιδίωμα, το centimorgan είναι περίπου 1 εκατομμύριο ζεύγη βάσεων.[15][16]
Μη φύσικο ζεύγος βάσεων (UBP)
[Επεξεργασία | επεξεργασία κώδικα]Ένα μη φύσικο ζεύγος βάσεων (unnatural base pair UBP) είναι μια σχεδιασμένη υπομονάδα (ή νουκλεοβάση) του DNA που δημιουργείται σε ένα εργαστήριο και δεν εμφανίζεται στη φύση. Έχουν περιγραφεί αλληλουχίες DNA που χρησιμοποιούν νεοδημιουργούμενες νουκλεοβάσεις για να σχηματίσουν ένα τρίτο ζεύγος βάσεων, επιπλέον των δύο ζευγών βάσεων που βρίσκονται στη φύση, A-T (αδενίνη – θυμίνη) και G-C (γουανίνη – κυτοσίνη). [17] Έχουν αναφερθεί ορισμένα νέα ζεύγη βάσεων που βασίζονται σε εναλλακτικούς δεσμούς υδρογόνου, υδρόφοβες αλληλεπιδράσεις και συντονισμό μετάλλων.[18][19][20][21] Το 1989 ο Steven Benner (τότε εργαζόταν στο Ομοσπονδιακό Ινστιτούτο Τεχνολογίας της Ζυρίχης) και η ομάδα του ηγήθηκαν με τροποποιημένες μορφές κυτοσίνης και γουανίνης σε μόρια DNA "in vitro".[22] Τα νουκλεοτίδια, τα οποία κωδικοποιούσαν το RNA και τις πρωτεΐνες, αντιγράφηκαν επιτυχώς "in vitro". Από τότε, η ομάδα του Benner προσπαθεί να κατασκευάσει κύτταρα που μπορούν να φτιάξουν ξένες βάσεις από την αρχή, αποφεύγοντας την ανάγκη για πρώτη ύλη.[23] Το 2002, η ομάδα του Ichiro Hirao στην Ιαπωνία ανέπτυξε ένα αφύσικο ζεύγος βάσεων μεταξύ 2-αμινο-8-(2-θειενυλ)πουρίνης και πυριδίνης-2-όνης που λειτουργεί στη μεταγραφή και τη μετάφραση, για τη συγκεκριμένη τοποθεσία ενσωμάτωσης μη τυπικών αμινοξέων σε πρωτεΐνες.[24] Το 2006, δημιούργησαν την 7-(2-θειενυλ)ιμιδαζο[4,5-b]πυριδίνη (Ds) και την πυρρόλη-2-καρβαλδεΰδη (Pa) ως τρίτο ζεύγος βάσεων για αντιγραφή και μεταγραφή.[25] Στη συνέχεια, το Ds και η 4-[3-(6-αμινοεξαναμιδο)-1-προπυνυλ]-2-νιτροπυρρόλη (Px) ανακαλύφθηκαν ως ζεύγος υψηλής πιστότητας στην ενίσχυση PCR.[26][27] Το 2013, εφάρμοσαν το ζεύγος Ds-Px στη δημιουργία απταμερών DNA με επιλογή in vitro (SELEX) και απέδειξαν ότι η επέκταση του γενετικού αλφαβήτου αυξάνει σημαντικά τις συγγένειες απταμερών DNA με πρωτεΐνες-στόχους.[28] Το 2012, μια ομάδα Αμερικανών επιστημόνων με επικεφαλής τον Floyd Romesberg, χημικό βιολόγο στο Scripps Research Institute στο Σαν Ντιέγκο της Καλιφόρνια, δημοσίευσε ότι η ομάδα του σχεδίασε ένα αφύσικο ζεύγος βάσεων (UBP).[20] Τα δύο νέα τεχνητά νουκλεοτίδια ή Αφύσικο Ζεύγος Βάσεων (UBP) ονομάστηκαν d5SICS και dNaM. Πιο τεχνικά, αυτά τα τεχνητά νουκλεοτίδια που φέρουν υδρόφοβες νουκλεοβάσεις διαθέτουν δύο συντηγμένους αρωματικούς δακτυλίους που σχηματίζουν ένα σύμπλοκο (d5SICS–dNaM) ή ζεύγος βάσεων στο DNA.[23][29] Η ομάδα του σχεδίασε μια ποικιλία προτύπων "in vitro" ή σε "δοκιμαστικό σωλήνα" που περιείχαν το αφύσικο ζεύγος βάσεων και επιβεβαίωσαν ότι αναπαράχθηκε αποτελεσματικά με υψηλή πιστότητα σχεδόν σε όλα τα πλαίσια ακολουθίας χρησιμοποιώντας τις σύγχρονες τυπικές τεχνικές "in vitro" , συγκεκριμένα ενίσχυση DNA με PCR και εφαρμογές που βασίζονται σε PCR.[20] Τα αποτελέσματά τους δείχνουν ότι για εφαρμογές που βασίζονται σε PCR και PCR, το ζεύγος μη φυσικών βάσεων d5SICS–dNaM είναι λειτουργικά ισοδύναμο με ένα φυσικό ζεύγος βάσεων και όταν συνδυάζεται με τα άλλα δύο ζεύγη φυσικών βάσεων που χρησιμοποιούνται από όλους τους οργανισμούς, A–T και G–C , παρέχουν ένα πλήρως λειτουργικό και διευρυμένο γενετικό αλφάβητο έξι γραμμάτων.[29] Το 2014 η ίδια ομάδα από το Ερευνητικό Ινστιτούτο Scripps ανέφερε ότι συνέθεσε ένα τμήμα κυκλικού DNA γνωστό ως πλασμίδιο που περιέχει φυσικά ζεύγη βάσεων T-A και C-G μαζί με το καλύτερο UBP του εργαστηρίου της Romesberg που το είχε σχεδιάσει και εισαγάγει στα κύτταρα του κοινού βακτηρίου E. coli που αναπαρήγαγε με επιτυχία τα μη φύσικα ζεύγη βάσεων σε πολλές γενιές.[17] Η επιμόλυνση δεν εμπόδισε την ανάπτυξη του E. coli και δεν έδειξε σημάδια απώλειας των μη φυσικών ζευγών βάσεων λόγω των φυσικών μηχανισμών [[ε[ιδιόρθωση του DNA|επιδιόρθωσης του DNA]]. Αυτό είναι το πρώτο γνωστό παράδειγμα ζωντανού οργανισμού που περνά κατά μήκος ενός διευρυμένου γενετικού κώδικα στις επόμενες γενιές.[29][30] Ο Romesberg είπε ότι αυτός και οι συνάδελφοί του δημιούργησαν 300 παραλλαγές για να βελτιώσουν το σχεδιασμό των νουκλεοτιδίων που θα ήταν αρκετά σταθερά και θα αναπαράγονταν τόσο εύκολα όσο τα φυσικά όταν διαιρούνται τα κύτταρα. Αυτό επιτεύχθηκε εν μέρει με την προσθήκη ενός υποστηρικτικού γονιδίου φυκιών που εκφράζει έναν μεταφορέα τριφωσφορικού νουκλεοτιδίου που εισάγει αποτελεσματικά τα τριφωσφορικά τόσο του d5SICSTP όσο και του dNaMTP στα βακτήρια E. coli.[29] Στη συνέχεια, οι φυσικές βακτηριακές οδοί αντιγραφής τα χρησιμοποίησαν για να αντιγράψουν με ακρίβεια ένα πλασμίδιο που περιέχει d5SICS–dNaM. Άλλοι ερευνητές εξεπλάγησαν που τα βακτήρια αντ[εγραψαν αυτές τις ανθρωπογενείς υπομονάδες DNA.[31] Η επιτυχής ενσωμάτωση ενός τρίτου ζεύγους βάσεων είναι μια σημαντική ανακάλυψη προς τον στόχο της μεγάλης επέκτασης του αριθμού των αμινοξέων που μπορούν να κωδικοποιηθούν από το DNA, από τα υπάρχοντα 20 αμινοξέα σε ένα θεωρητικά πιθανό 172, επεκτείνοντας έτσι το δυνατότητα για ζωντανούς οργανισμούς να παράγουν νέες πρωτεΐνες.[17] Οι τεχνητές χορδές του DNA δεν κωδικοποιούν τίποτα ακόμα, αλλά οι επιστήμονες εικάζουν ότι θα μπορούσαν να σχεδιαστούν για να παράγουν νέες πρωτεΐνες που θα μπορούσαν να έχουν βιομηχανική ή φαρμακευτική χρήση.[32] Οι ειδικοί είπαν ότι το συνθετικό DNA που ενσωματώνει το μη φύσικο ζεύγος βάσεων αυξάνει την πιθανότητα μορφών ζωής που βασίζονται σε διαφορετικό κώδικα DNA.[31][32]
Μη κανονική σύζευξη βάσεων
[Επεξεργασία | επεξεργασία κώδικα]Εκτός από την κανονική σύζευξη, ορισμένες συνθήκες μπορούν επίσης να ευνοήσουν το ζευγάρωμα βάσεων με εναλλακτικό προσανατολισμό βάσης και αριθμό και γεωμετρία των δεσμών υδρογόνου. Αυτά τα ζεύγη συνοδεύονται από αλλαγές στο τοπικό σχήμα σκελετού. Το πιο κοινό από αυτά είναι το ταλαντευόμενο ζεύγος βάσεων (wobble base pairing) που συμβαίνει μεταξύ tRNA και mRNA στην τρίτη θέση βάσης πολλών κωδονίων κατά τη διάρκεια της μεταγραφής[34] και κατά τη φόρτιση των tRNA από ορισμένες συνθετάσες tRNA.[35] Έχουν επίσης παρατηρηθεί στις δευτεροταγείς δομές ορισμένων αλληλουχιών RNA.[36] Επιπλέον, το ζεύγος βάσεων Hoogsteen (συνήθως γράφεται ως A•U/T και G•C) μπορεί να υπάρχει σε ορισμένες αλληλουχίες DNA (π.χ. δινουκλεοτίδια CA και TA) σε δυναμική ισορροπία με την τυπική σύζευξη Watson-Crick.[33] Έχουν επίσης παρατηρηθεί σε ορισμένα σύμπλοκα πρωτεΐνης-DNA.[37] Εκτός από αυτά τα εναλλακτικά ζεύγη βάσεων, ένα ευρύ φάσμα δεσμών υδρογόνου βάσης-βάσης παρατηρείται στη δευτεροταγή και τριτοταγή δομή RNA.[38] Αυτοί οι δεσμοί είναι συχνά απαραίτητοι για το ακριβές, πολύπλοκο σχήμα ενός RNA, καθώς και για τη σύνδεσή του με τους εταίρους αλληλεπίδρασης.[38]
Παραπομπές
[Επεξεργασία | επεξεργασία κώδικα]- ↑ «The stereochemistry of deoxyribonucleic acid. II. Hydrogen-bonded pairs of bases» (στα αγγλικά). Acta Crystallographica 12 (1): 66–71. 10 January 1959. doi:. ISSN 0365-110X. https://scripts.iucr.org/cgi-bin/paper?S0365110X59000160.
- ↑ Zhurkin VB, Tolstorukov MY, Xu F, Colasanti AV, Olson WK (2005). «Sequence-Dependent Variability of B-DNA». DNA Conformation and Transcription. σελίδες 18–34. doi:10.1007/0-387-29148-2_2. ISBN 978-0-387-25579-8.
- ↑ Moran LA (24 Μαρτίου 2011). «The total size of the human genome is very likely to be ~3,200 Mb». Sandwalk.blogspot.com. Ανακτήθηκε στις 16 Ιουλίου 2012.
- ↑ «The finished length of the human genome is 2.86 Gb». Strategicgenomics.com. 12 Ιουνίου 2006. Ανακτήθηκε στις 16 Ιουλίου 2012.
- ↑ «One copy of the human genome consists of approximately 3 billion base pairs of DNA». National Human Genome Research Institute. 24 Αυγούστου 2024.
- ↑ «Finishing the euchromatic sequence of the human genome». Nature 431 (7011): 931–945. October 2004. doi:. PMID 15496913. Bibcode: 2004Natur.431..931H.
- ↑ «Organization of the ribosomal RNA genes of Dictyostelium discoideum: mapping of the nontranscribed spacer regions». Cell 9 (4 Pt 1): 605–613. December 1976. doi:. PMID 1034500.
- ↑ «Counting All the DNA on Earth». The New York Times (New York). 18 July 2015. ISSN 0362-4331. Αρχειοθετήθηκε από το πρωτότυπο στις 2022-01-01. https://ghostarchive.org/archive/20220101/https://www.nytimes.com/2015/07/21/science/counting-all-the-dna-on-earth.html. Ανακτήθηκε στις 2015-07-18.
- ↑ «The Biosphere: Diversity of Life». Aspen Global Change Institute. Basalt, CO. Αρχειοθετήθηκε από το πρωτότυπο στις 10 Νοεμβρίου 2014. Ανακτήθηκε στις 19 Ιουλίου 2015.
- ↑ «Base-stacking and base-pairing contributions into thermal stability of the DNA double helix». Nucleic Acids Research 34 (2): 564–574. 2006-01-30. doi:. PMID 16449200.
- ↑ «Enzymatic synthesis of deoxyribonucleic acid. X. Influence of bromouracil substitutions on replication». Proceedings of the National Academy of Sciences of the United States of America 48 (3): 449–455. March 1962. doi:. PMID 13922323.
- ↑ Krebs JE, Goldstein ES, Kilpatrick ST, Lewin B (2018). «Genes are DNA and Encode RNAs and Polypeptides». Lewin's genes XII (12th έκδοση). Burlington, Mass: Jones & Bartlett Learning. σελ. 12. ISBN 978-1-284-10449-3.
Each mutagenic event in the presence of an acridine results in the addition or removal of a single base pair.
- ↑ «Strand discrimination in DNA mismatch repair». DNA Repair 105: 103161. September 2021. doi:. PMID 34171627.
- ↑ Alberts B, Johnson A, Lewis J, Morgan D, Raff M, Roberts K, Walter P (Δεκεμβρίου 2014). Molecular Biology of the Cell (6th έκδοση). New York/Abingdon: Garland Science, Taylor & Francis Group. σελ. 177. ISBN 978-0-8153-4432-2.
- ↑ «NIH ORDR – Glossary – C». Rarediseases.info.nih.gov. Αρχειοθετήθηκε από το πρωτότυπο στις 17 Ιουλίου 2012. Ανακτήθηκε στις 16 Ιουλίου 2012.
- ↑ Scott MP, Matsudaira P, Lodish H, Darnell J, Zipursky L, Kaiser CA, Berk A, Krieger M (2004). Molecular Cell Biology
 (Fifth έκδοση). San Francisco: W. H. Freeman. σελ. 396. ISBN 978-0-7167-4366-8.
(Fifth έκδοση). San Francisco: W. H. Freeman. σελ. 396. ISBN 978-0-7167-4366-8. ...in humans 1 centimorgan on average represents a distance of about 7.5x105 base pairs.
- ↑ 17,0 17,1 17,2 «Life engineered with expanded genetic code». San Diego Union Tribune. May 8, 2014. Αρχειοθετήθηκε από το πρωτότυπο στις 9 May 2014. https://web.archive.org/web/20140509001048/http://www.utsandiego.com/news/2014/may/08/tp-life-engineered-with-expanded-genetic-code/. Ανακτήθηκε στις 8 May 2014.
- ↑ «Amplification, mutation, and sequencing of a six-letter synthetic genetic system». Journal of the American Chemical Society 133 (38): 15105–15112. September 2011. doi:. PMID 21842904.
- ↑ «Highly specific unnatural base pair systems as a third base pair for PCR amplification». Nucleic Acids Research 40 (6): 2793–2806. March 2012. doi:. PMID 22121213.
- ↑ 20,0 20,1 20,2 «Efficient and sequence-independent replication of DNA containing a third base pair establishes a functional six-letter genetic alphabet». Proceedings of the National Academy of Sciences of the United States of America 109 (30): 12005–12010. July 2012. doi:. PMID 22773812. Bibcode: 2012PNAS..10912005M.
- ↑ «Artificial DNA Base Pairing Mediated by Diverse Metal Ions» (στα αγγλικά). Chemistry Letters 46 (5): 622–633. 2017-05-05. doi:. ISSN 0366-7022.
- ↑ «Enzymatic incorporation of a new base pair into DNA and RNA». J. Am. Chem. Soc. 111 (21): 8322–8323. 1989. doi:.
- ↑ 23,0 23,1 «Scientists Create First Living Organism With 'Artificial' DNA». Nature News (Huffington Post). May 7, 2014. http://www.huffingtonpost.com/2014/05/07/living-organism-artificial-dna_n_5283095.html. Ανακτήθηκε στις 8 May 2014.
- ↑ «An unnatural base pair for incorporating amino acid analogs into proteins». Nature Biotechnology 20 (2): 177–182. February 2002. doi:. PMID 11821864.
- ↑ «An unnatural hydrophobic base pair system: site-specific incorporation of nucleotide analogs into DNA and RNA». Nature Methods 3 (9): 729–735. September 2006. doi:. PMID 16929319.
- ↑ «An unnatural base pair system for efficient PCR amplification and functionalization of DNA molecules». Nucleic Acids Research 37 (2): e14. February 2009. doi:. PMID 19073696.
- ↑ «Highly specific unnatural base pair systems as a third base pair for PCR amplification». Nucleic Acids Research 40 (6): 2793–2806. March 2012. doi:. PMID 22121213.
- ↑ «Generation of high-affinity DNA aptamers using an expanded genetic alphabet». Nature Biotechnology 31 (5): 453–457. May 2013. doi:. PMID 23563318.
- ↑ 29,0 29,1 29,2 29,3 «A semi-synthetic organism with an expanded genetic alphabet». Nature 509 (7500): 385–388. May 2014. doi:. PMID 24805238. Bibcode: 2014Natur.509..385M.
- ↑ «First life forms to pass on artificial DNA engineered by US scientists». The Guardian. May 7, 2014. https://www.theguardian.com/world/2014/may/07/living-organism-pass-down-artificial-dna-us-scientists. Ανακτήθηκε στις 8 May 2014.
- ↑ 31,0 31,1 «Scientists create first living organism containing artificial DNA». The Wall Street Journal (Fox News). May 8, 2014. https://www.foxnews.com/health/scientists-create-first-living-organism-containing-artificial-dna/. Ανακτήθηκε στις 8 May 2014.
- ↑ 32,0 32,1 «Scientists Add Letters to DNA's Alphabet, Raising Hope and Fear». New York Times. May 7, 2014. https://www.nytimes.com/2014/05/08/business/researchers-report-breakthrough-in-creating-artificial-genetic-code.html?hpw&rref=business&_r=0. Ανακτήθηκε στις 8 May 2014.
- ↑ 33,0 33,1 «Transient Hoogsteen base pairs in canonical duplex DNA». Nature 470 (7335): 498–502. February 2011. doi:. PMID 21270796. Bibcode: 2011Natur.470..498N.
- ↑ «Structure of a purine-purine wobble base pair in the decoding center of the ribosome». Nature Structural & Molecular Biology 11 (12): 1251–1252. December 2004. doi:. PMID 15558050.
- ↑ «Structural biology: wobble puts RNA on target». Nature 510 (7506): 480–481. June 2014. doi:. PMID 24919145.
- ↑ «A novel form of RNA double helix based on G•U and C•A+ wobble base pairing». RNA 24 (2): 209–218. February 2018. doi:. PMID 29122970.
- ↑ «A Hoogsteen base pair embedded in undistorted B-DNA». Nucleic Acids Research 30 (23): 5244–5252. December 2002. doi:. PMID 12466549.
- ↑ 38,0 38,1 «Analysis of RNA motifs». Current Opinion in Structural Biology 13 (3): 300–308. June 2003. doi:. PMID 12831880.
Περαιτέρω μελέτη
[Επεξεργασία | επεξεργασία κώδικα]- Watson JD, Baker TA, Bell SP, Gann A, Levine M, Losick R (2004). Molecular Biology of the Gene (5th έκδοση). Pearson Benjamin Cummings: CSHL Press. (See esp. ch. 6 and 9)
- Sigel A, Sigel H, Sigel RK, επιμ. (2012). Interplay between Metal Ions and Nucleic Acids. Metal Ions in Life Sciences. 10. Springer. doi:10.1007/978-94-007-2172-2. ISBN 978-9-4007-2171-5. Unknown parameter
|s2cid=ignored (βοήθεια) - Clever GH, Shionoya M (2012). «Alternative DNA Base Pairing through Metal Coordination». Interplay between Metal Ions and Nucleic Acids. Metal Ions in Life Sciences. 10. σελίδες 269–294. doi:10.1007/978-94-007-2172-2_10. ISBN 978-94-007-2171-5. PMID 22210343.
- Megger DA, Megger N, Mueller J (2012). «Metal-Mediated Base Pairs in Nucleic Acids with Purine- and Pyrimidine-Derived Nucleosides». Interplay between Metal Ions and Nucleic Acids. Metal Ions in Life Sciences. 10. σελίδες 295–317. doi:10.1007/978-94-007-2172-2_11. ISBN 978-94-007-2171-5. PMID 22210344.