Μεταγραφή (βιολογία)
Μεταγραφή είναι η διαδικασία αντιγραφής ενός τμήματος του DNA στο RNA με σκοπό την γονιδιακή έκφραση. Ορισμένα τμήματα του DNA μεταγράφονται σε μόρια RNA που μπορούν να κωδικοποιήσουν πρωτεΐνες, που ονομάζονται αγγελιοφόρο RNA (mRNA). Άλλα τμήματα του DNA μεταγράφονται σε μόρια RNA που ονομάζονται μη κωδικοποιητικά RNA (non-coding RNAs, ncRNAs). Τόσο το DNA όσο και το RNA είναι νουκλεϊκά οξέα, τα οποία χρησιμοποιούν ζεύγη βάσεων των νουκλεοτιδίων ως συμπληρωματική γλώσσα. Κατά τη μεταγραφή, μια αλληλουχία DNA διαβάζεται από μία RNA πολυμεράση, που παράγει έναν συμπληρωματικό, αντιπαράλληλο κλώνο RNA που ονομάζεται πρωτογενές μεταγράφημα (primary transcript). Στην ιολογία, ο όρος μεταγραφή χρησιμοποιείται όταν αναφέρεται στη σύνθεση mRNA από ένα μόριο ιικού RNA. Το γονιδίωμα πολλών ιών RNA[α] αποτελείται από αρνητικό νοηματικό RNA που λειτουργεί ως πρότυπο για νοηματικά RNA αγγελιαφόρου ιού - ένα απαραίτητο βήμα στη σύνθεση ιικών πρωτεϊνών που απαιτούνται για ιική αντιγραφή. Αυτή η διαδικασία καταλύεται από μια ιική πολυμεράση RNA εξαρτώμενη από RNA.[1]
Υπόβαθρο
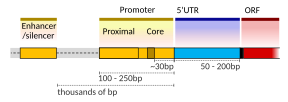
[Επεξεργασία | επεξεργασία κώδικα]Μια μονάδα μεταγραφής DNA που κωδικοποιεί μια πρωτεΐνη μπορεί να περιέχει τόσο μια κωδικοποιητική αλληλουχία, η οποία θα μεταφραστεί στην πρωτεΐνη, όσο και ρυθμιστικές αλληλουχίες, που κατευθύνουν και ρυθμίζουν τη σύνθεση αυτής της πρωτεΐνης. Η ρυθμιστική αλληλουχία πριν την ανάντη από την κωδικοποιητική αλληλουχία ονομάζεται 5' αμετάφραστη περιοχή (five prime untranslated region, 5'UTR). η ακολουθία μετά την κατάντη από την κωδικοποιητική ακολουθία ονομάζεται 3' αμετάφραστη περιοχή (three prime untranslated region, 3'UTR).[2] Σε αντίθεση με την αντιγραφή του DNA, η μεταγραφή έχει ως αποτέλεσμα ένα συμπλήρωμα RNA που περιλαμβάνει το νουκλεοτίδιο ουρακίλη (U) σε όλες τις περιπτώσεις όπου η θυμίνη (Τ) θα είχε εμφανιστεί σε ένα συμπλήρωμα DNA.[3] Μόνο ένας από τους δύο κλώνους DNA χρησιμεύει ως πρότυπο για τη μεταγραφή. Ο αντινοηματικός κλώνος του DNA διαβάζεται από την RNA πολυμεράση από το 3' άκρο προς το 5' άκρο κατά τη διάρκεια της μεταγραφής (3' → 5'). Το συμπληρωματικό RNA δημιουργείται προς την αντίθετη κατεύθυνση, προς την κατεύθυνση 5' → 3', ταιριάζοντας με την αλληλουχία του νοηματικού κλώνου εκτός από την εναλλαγή της ουρακίλης για τη θυμίνη. Αυτή η κατευθυντικότητα οφείλεται στο ότι η RNA πολυμεράση μπορεί να προσθέσει νουκλεοτίδια μόνο στο 3' άκρο της αναπτυσσόμενης αλυσίδας mRNA. Αυτή η χρήση μόνο του 3' → 5' κλώνου DNA εξαλείφει την ανάγκη για τα θραύσματα του Οκαζάκι που φαίνονται στην αντιγραφή του DNA.[2] Αυτό αφαιρεί επίσης την ανάγκη για ένα RNA εκκινητή για την έναρξη της σύνθεσης RNA, όπως συμβαίνει στην αντιγραφή του DNA. Ο νοηματικός κλώνος του DNA ονομάζεται κωδικοποιητικός κλώνος, επειδή η αλληλουχία του είναι η ίδια με το νεοδημιουργημένο μεταγράφημα RNA (εκτός από την υποκατάσταση της ουρακίλης αντί της θυμίνης). Αυτός είναι ο κλώνος που χρησιμοποιείται κατά σύμβαση όταν παρουσιάζεται μια αλληλουχία DNA.[4] Η μεταγραφή έχει ορισμένους μηχανισμούς διόρθωσης, αλλά είναι λιγότεροι και λιγότερο αποτελεσματικοί από τους ελέγχους για την αντιγραφή του DNA. Ως αποτέλεσμα, η μεταγραφή έχει χαμηλότερη πιστότητα αντιγραφής από την αντιγραφή του DNA.[5]
Βασικά βήματα
[Επεξεργασία | επεξεργασία κώδικα]Η μεταγραφή χωρίζεται σε έναρξη, διαφυγή προαγωγέα, επιμήκυνση και τερματισμός.[6]
Ρύθμιση για μεταγραφή
[Επεξεργασία | επεξεργασία κώδικα]Ενισχυτές, παράγοντες μεταγραφής, σύμπλοκο μεσολαβητή και βρόχοι DNA στη μεταγραφή θηλαστικών
[Επεξεργασία | επεξεργασία κώδικα]
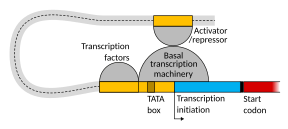
Η ρύθμιση για τη μεταγραφή σε θηλαστικά ρυθμίζεται από πολλά cis ρυθμιστικά στοιχεία, συμπεριλαμβανομένων των εγγύς στοιχείων του προαγωγέα που βρίσκονται κοντά στις θέσεις έναρξης της μεταγραφής των γονιδίων. Οι βασικοί προαγωγείς σε συνδυασμό με τους γενικούς παράγοντες μεταγραφής είναι επαρκείς για να κατευθύνουν την έναρξη της μεταγραφής, αλλά γενικά έχουν χαμηλή βασική δραστηριότητα.[7] Άλλες σημαντικές ρυθμιστικές μονάδες cis εντοπίζονται σε περιοχές DNA που είναι απομακρυσμένες από τις θέσεις έναρξης της μεταγραφής. Αυτές περιλαμβάνουν ενισχυτές, αποσιωπητές, μονωτές και στοιχεία πρόσδεσης.[8] Μεταξύ αυτών των πολλών στοιχείων, οι ενισχυτές και οι σχετικοί παράγοντες μεταγραφής έχουν πρωταγωνιστικό ρόλο στην έναρξη της γονιδιακής μεταγραφής.[9] Ένας ενισχυτής εντοπισμένος σε μια περιοχή DNA μακριά από τον προαγωγέα ενός γονιδίου μπορεί να έχει πολύ μεγάλη επίδραση στη μεταγραφή του γονιδίου, με ορισμένα γονίδια να υφίστανται έως και 100 φορές αυξημένη μεταγραφή λόγω ενός ενεργοποιημένου ενισχυτή.[10]
Οι ενισχυτές είναι περιοχές του γονιδιώματος που είναι κύρια γονιδιακά ρυθμιστικά στοιχεία. Οι ενισχυτές ελέγχουν προγράμματα μεταγραφής γονιδίων ειδικά για τον κυτταρικό τύπο, τις περισσότερες φορές κάνοντας βρόχους σε μεγάλες αποστάσεις για να έρθουν σε φυσική εγγύτητα με τους προαγωγείς των γονιδίων-στόχων τους.[11] Ενώ υπάρχουν εκατοντάδες χιλιάδες περιοχές DNA ενισχυτών,[12] για έναν συγκεκριμένο τύπο ιστού μόνο ειδικοί ενισχυτές έρχονται σε εγγύτητα με τους προαγωγούς που ρυθμίζουν. Σε μια μελέτη των νευρώνων του φλοιού του εγκεφάλου, βρέθηκαν 24.937 βρόχοι, που φέρνουν ενισχυτές στους προαγωγείς-στόχους τους.[10] Πολλοί ενισχυτές, ο καθένας συχνά σε δεκάδες ή εκατοντάδες χιλιάδες νουκλεοτίδια απομακρυσμένος από τα γονίδια-στόχους τους, συνδέονται με τους προαγωγείς του γονιδίου στόχου τους και μπορούν να συντονιστούν μεταξύ τους για τον έλεγχο της μεταγραφής του κοινού γονιδίου στόχου τους.[11] Η σχηματική απεικόνιση σε αυτό το τμήμα δείχνει έναν ενισχυτή να κάνει βρόχο για να έρθει σε στενή φυσική εγγύτητα με τον προαγωγέα ενός γονιδίου στόχου. Ο βρόχος σταθεροποιείται από ένα διμερές μιας πρωτεΐνης σύνδεσης (π.χ. διμερές του CTCF ή YY1), με ένα μέλος του διμερούς αγκυρωμένο στο μοτίβο δέσμευσής του στον ενισχυτή και το άλλο μέλος αγκυρωμένο στη σύνδεσή του μοτίβου στον προαγωγέα (που αντιπροσωπεύεται από τα κόκκινα ζιγκ-ζαγκ στην εικόνα).[13] Υπάρχουν αρκετοί μεταγραφικοί παράγοντες ειδικής λειτουργίας των κυττάρων (περίπου 1.600 μεταγραφικοί παράγοντες σε ένα ανθρώπινο κύτταρο [14]) που συνδέονται γενικά με συγκεκριμένα μοτίβα σε έναν ενισχυτή [15] Και ένας μικρός συνδυασμός αυτών των μεταγραφικών παραγόντων που συνδέονται με τον ενισχυτή, όταν έρχονται κοντά σε έναν προαγωγέα μέσω βρόχου DNA, διέπουν το επίπεδο μεταγραφής του γονιδίου στόχου. Ο διαμεσολαβητής (ένα σύμπλοκο που αποτελείται συνήθως από περίπου 26 πρωτεΐνες σε μια δομή αλληλεπίδρασης) επικοινωνεί με ρυθμιστικά σήματα με τους μεταγραφικούς παράγοντες που δεσμεύονται στο DNA του ενισχυτή απευθείας στο ένζυμο RNA πολυμεράσης II (pol II) που είναι συνδεδεμένο με τον προαγωγέα.[16] Οι ενισχυτές, όταν είναι ενεργοί, μεταγράφονται γενικά και από τους δύο κλώνους του DNA με τις RNA πολυμεράσες που δρουν σε δύο διαφορετικές κατευθύνσεις, παράγοντας δύο ενισχυτικά RNAs (enhancer RNAs, eRNAs) όπως απεικονίζεται στο σχήμα.[17] Ένας ανενεργός ενισχυτής μπορεί να δεσμεύεται από έναν ανενεργό παράγοντα μεταγραφής. Η φωσφορυλίωση του μεταγραφικού παράγοντα μπορεί να τον ενεργοποιήσει και αυτός ο ενεργοποιημένος παράγοντας μεταγραφής μπορεί στη συνέχεια να ενεργοποιήσει τον ενισχυτή στον οποίο είναι συνδεδεμένος (βλ. το μικρό κόκκινο αστέρι που αντιπροσωπεύει τη φωσφορυλίωση του μεταγραφικού παράγοντα που συνδέεται με τον ενισχυτή στην εικόνα).[18] Ένας ενεργοποιημένος ενισχυτής ξεκινά τη μεταγραφή του RNA του πριν ενεργοποιήσει τη μεταγραφή του αγγελιαφόρου RNA από το γονίδιο στόχο του.[19]
Μεθυλίωση και απομεθυλίωση νησίδας CpG
[Επεξεργασία | επεξεργασία κώδικα]
Η ρύθμιση της μεταγραφής στο 60% περίπου των προαγωγέων ελέγχεται επίσης με μεθυλίωση κυτοσινών εντός των δινουκλεοτιδίων CpG (όπου η 5' κυτοσίνη ακολουθείται από 3' γουανίνη ή θέσεις CpG). Η 5-μεθυλκυτοσίνη (5-mC) είναι μια μεθυλιωμένη μορφή της βάσης κυτοσίνη του DNA (βλ. Εικόνα). Η 5-mC είναι ένας δείκτης επιγενετικής που βρίσκεται κυρίως στις θέσεις CpG. Περίπου 28 εκατομμύρια δινουκλεοτίδια CpG εμφανίζονται στο ανθρώπινο γονιδίωμα.[20] Στους περισσότερους ιστούς θηλαστικών, κατά μέσο όρο, το 70% έως 80% των κυτοσινών CpG είναι μεθυλιωμένες (σχηματίζοντας 5-methylCpG ή 5-mCpG).[21] Ωστόσο, οι μη μεθυλιωμένες κυτοσίνες εντός των αλληλουχιών 5'κυτοσίνης-γουανίνης 3' εμφανίζονται συχνά σε ομάδες, που ονομάζονται νησίδες CpG, σε ενεργούς προαγωγείς. Περίπου το 60% των αλληλουχιών προαγωγέα έχουν νησίδα CpG, ενώ μόνο το 6% περίπου των αλληλουχιών ενισχυτών έχουν νησίδα CpG.[22] Οι νησίδες CpG αποτελούν ρυθμιστικές αλληλουχίες, καθώς εάν οι νησίδες CpG μεθυλιωθούν στον προαγωγέα ενός γονιδίου, αυτό μπορεί να μειώσει ή να αποσιωπήσει τη μεταγραφή του γονιδίου.[23] Η μεθυλίωση του DNA ρυθμίζει τη γονιδιακή μεταγραφή μέσω αλληλεπίδρασης με πρωτεΐνες του τομέα δέσμευσης μεθυλίου (methyl binding domain, MBD), όπως MeCP2, MBD1 και MBD2. Αυτές οι πρωτεΐνες MBD συνδέονται πιο ισχυρά με υψηλά μεθυλιωμένες νησίδες CpG.[24] Αυτές οι πρωτεΐνες MBD έχουν τόσο μια περιοχή δέσμευσης μεθυλ-CpG όσο και μια περιοχή καταστολής μεταγραφής.[24] Συνδέονται με μεθυλιωμένο DNA και καθοδηγούν ή κατευθύνουν σύμπλοκα πρωτεϊνών με δράση αναδιαμόρφωσης χρωματίνης ή/και τροποποιητικής ιστόνης σε μεθυλιωμένες νησίδες CpG. Οι πρωτεΐνες MBD καταστέλλουν γενικά την τοπική χρωματίνη, όπως καταλύοντας την εισαγωγή των κατασταλτικών σημαδιών ιστόνης ή δημιουργώντας ένα συνολικό κατασταλτικό περιβάλλον χρωματίνης μέσω αναδιαμόρφωσης νουκλεοσώματος και αναδιοργάνωσης της χρωματίνης.[24]

Όπως σημειώθηκε στην προηγούμενη ενότητα, οι παράγοντες μεταγραφής είναι πρωτεΐνες που συνδέονται με συγκεκριμένες αλληλουχίες DNA προκειμένου να ρυθμίσουν την έκφραση ενός γονιδίου. Η αλληλουχία δέσμευσης για έναν παράγοντα μεταγραφής στο DNA έχει συνήθως μήκος περίπου 10 ή 11 νουκλεοτιδίων. Όπως συνοψίστηκε το 2009, οι Vaquerizas κ.α. υποδεικνύουν ότι υπάρχουν περίπου 1.400 διαφορετικοί παράγοντες μεταγραφής που κωδικοποιούνται στο ανθρώπινο γονιδίωμα από γονίδια που αποτελούν περίπου το 6% όλων των γονιδίων που κωδικοποιούν ανθρώπινη πρωτεΐνη.[25] Περίπου το 94% των θέσεων δέσμευσης μεταγραφικού παράγοντα (transcription factor binding sites, TFBS) που σχετίζονται με γονίδια που ανταποκρίνονται στο σήμα εμφανίζονται σε ενισχυτές, ενώ μόνο περίπου το 6% αυτών των TFBS εμφανίζονται σε προαγωγείς.[15] Η πρωτεΐνη EGR1 είναι ένας ιδιαίτερος μεταγραφικός παράγοντας που είναι σημαντικός για τη ρύθμιση της μεθυλίωσης των νησίδων CpG. Μια θέση δέσμευσης μεταγραφικού παράγοντα EGR1 εντοπίζεται συχνά σε αλληλουχίες ενισχυτή ή προαγωγέα.[26] Υπάρχουν περίπου 12.000 θέσεις δέσμευσης για το EGR1 στο γονιδίωμα των θηλαστικών και περίπου οι μισές θέσεις δέσμευσης EGR1 βρίσκονται σε προαγωγείς και οι μισές σε ενισχυτές.[26] Η δέσμευση του EGR1 στη θέση δέσμευσης του DNA στόχου είναι μη ευαίσθητη στη μεθυλίωση της κυτοσίνης στο DNA.[26] Ενώ μόνο μικρές ποσότητες πρωτεΐνης μεταγραφικού παράγοντα EGR1 είναι ανιχνεύσιμες σε κύτταρα που δεν διεγείρονται, η μετάφραση του γονιδίου EGR1 σε πρωτεΐνη μία ώρα μετά τη διέγερση είναι δραστικά αυξημένη.[27] Η παραγωγή πρωτεϊνών του μεταγραφικού παράγοντα EGR1, σε διάφορους τύπους κυττάρων, μπορεί να διεγερθεί από αυξητικούς παράγοντες, νευροδιαβιβαστές, ορμόνες, άγχος και τραυματισμούς.[27] Στον εγκέφαλο, όταν ενεργοποιούνται οι νευρώνες, οι πρωτεΐνες EGR1 ρυθμίζονται προς τα πάνω και συνδέονται με (στρατολογούν) τα προϋπάρχοντα ένζυμα TET1 που παράγονται σε υψηλές ποσότητες στους νευρώνες. Τα ένζυμα ΤΕΤ μπορούν να καταλύσουν την απομεθυλίωση της 5-μεθυλκυτοσίνης. Όταν οι μεταγραφικοί παράγοντες EGR1 φέρνουν τα ένζυμα TET1 σε θέσεις δέσμευσης EGR1 σε προαγωγείς, τα ένζυμα TET μπορούν να απομεθυλιώσουν τις μεθυλιωμένες νησίδες CpG σε αυτούς τους προαγωγείς. Κατά την απομεθυλίωση, αυτοί οι προαγωγείς μπορούν στη συνέχεια να ξεκινήσουν τη μεταγραφή των γονιδίων-στόχων τους. Εκατοντάδες γονίδια σε νευρώνες εκφράζονται διαφορετικά μετά την ενεργοποίηση νευρώνων μέσω της στρατολόγησης EGR1 του TET1 σε μεθυλιωμένες ρυθμιστικές αλληλουχίες στους προαγωγείς τους.[26] Η μεθυλίωση των προαγωγέων μεταβάλλεται επίσης ως απόκριση στα σήματα. Οι τρεις μεθυλομεταφοράσες των θηλαστικών (DNMT1, DNMT3A και DNMT3B) καταλύουν την προσθήκη μεθυλομάδων σε κυτοσίνες στο DNA. Ενώ το DNMT1 είναι μεθυλομεταφοράση συντήρησης, το DNMT3A και το DNMT3B μπορούν να πραγματοποιήσουν νέες μεθυλιώσεις. Υπάρχουν επίσης δύο ισομορφές ματίσματος πρωτεϊνών που παράγονται από το γονίδιο DNMT3A: οι μεθυλομεταφοράσες του DNA DNMT3A1 και DNMT3A2.[28] Η ισομορφή ματίσματος DNMT3A2 συμπεριφέρεται όπως το προϊόν ενός κλασικού άμεσου-πρώιμου γονιδίου και, για παράδειγμα, παράγεται ισχυρά και παροδικά μετά από νευρωνική ενεργοποίηση.[29] Όπου η ισομορφή DNMT3A2 μεθυλομεταφοράσης DNA δεσμεύεται και προσθέτει μεθυλικές ομάδες στις κυτοσίνες φαίνεται να προσδιορίζεται από τροποποιήσεις ιστόνης μετά τη μετάφραση.[30][31][32] Από την άλλη πλευρά, η νευρική ενεργοποίηση προκαλεί αποικοδόμηση του DNMT3A1 που συνοδεύεται από μειωμένη μεθυλίωση τουλάχιστον ενός αξιολογημένου στοχευμένου προαγωγέα.[33]
Έναρξη
[Επεξεργασία | επεξεργασία κώδικα]Η μεταγραφή ξεκινά με την RNA πολυμεράση και έναν ή περισσότερους γενικούς μεταγραφικούς παράγοντες που συνδέονται με μια αλληλουχία DNA προαγωγέα για να σχηματίσουν ένα κλειστό σύμπλεγμα RNA πολυμεράσης-προαγωγέα. Στο κλειστό σύμπλεγμα, το DNA του προαγωγέα εξακολουθεί να είναι πλήρως δίκλωνο.[6] Η RNA πολυμεράση, υποβοηθούμενη από έναν ή περισσότερους γενικούς μεταγραφικούς παράγοντες, στη συνέχεια ξετυλίγει περίπου 14 ζεύγη βάσεων DNA για να σχηματίσει ένα ανοιχτό σύμπλεγμα RNA πολυμεράσης-προαγωγέα. Στο ανοιχτό σύμπλεγμα, το DNA του προαγωγέα είναι εν μέρει ξετυλιγμένο και μονόκλωνο. Το εκτεθειμένο, μονόκλωνο DNA αναφέρεται ως φυσαλίδα μεταγραφής (transcription bubble).[6] Η RNA πολυμεράση, υποβοηθούμενη από έναν ή περισσότερους γενικούς παράγοντες μεταγραφής, στη συνέχεια επιλέγει μια θέση έναρξης μεταγραφής στη φυσαλίδα μεταγραφής, δεσμεύεται σε ένα αρχικό τριφωσφορικό νουκλεοζίτη και ένα επεκτεινόμενο τριφωσφορικό νουκλεοζίτη (ή ένα σύντομο εκκινητή RNA και ένα επεκτεινόμενο NTP) συμπληρωματικό στην αλληλουχία θέσης έναρξης της μεταγραφής και καταλύει το σχηματισμό δεσμού για να δώσει ένα αρχικό προϊόν RNA.[6] Στα βακτήρια, το ολοένζυμο RNA πολυμεράσης αποτελείται από πέντε υπομονάδες: 2 α υπομονάδες, 1 β υπομονάδα, 1 β' υπομονάδα και 1 ω υπομονάδα. Στα βακτήρια, υπάρχει ένας γενικός παράγοντας μεταγραφής RNA γνωστός ως παράγοντας σίγμα. Το κεντρικό ένζυμο RNA πολυμεράσης συνδέεται με τον βακτηριακό γενικό παράγοντα μεταγραφής (σίγμα) για να σχηματίσει το ολοένζυμο πολυμεράσης RNA και στη συνέχεια συνδέεται με έναν προαγωγέα.[6] (Η RNA πολυμεράση ονομάζεται ολοένζυμο όταν η υπομονάδα σίγμα συνδέεται με το κύριο ένζυμο που αποτελείται από 2 α υπομονάδες, 1 β υπομονάδα, 1 β' υπομονάδα μόνο). Σε αντίθεση με τους ευκαρυώτες, το νουκλεοτίδιο έναρξης του εκκολαπτόμενου βακτηριακού mRNA δεν καλύπτεται με ένα τροποποιημένο νουκλεοτίδιο γουανίνης. Το νουκλεοτίδιο έναρξης των βακτηριακών μεταγραφών φέρει ένα 5' τριφωσφορικό (5'-PPP), το οποίο μπορεί να χρησιμοποιηθεί για χαρτογράφηση θέσεων έναρξης μεταγραφής σε όλο το γονιδίωμα.[35] Στα αρχαία και τους ευκαρυώτες, η RNA πολυμεράση περιέχει ομόλογες υπομονάδες σε καθεμία από τις πέντε υπομονάδες RNA πολυμεράσης στα βακτήρια και περιέχει επίσης επιπλέον υπομονάδες. Στα αρχαία και στους ευκαρυώτες, οι λειτουργίες του βακτηριακού γενικού μεταγραφικού παράγοντα σίγμα εκτελούνται από πολλαπλούς γενικούς μεταγραφικούς παράγοντες που συνεργάζονται.[6] Στα αρχαία, υπάρχουν τρεις γενικοί μεταγραφικοί παράγοντες: TBP, TFB και TFE. Στους ευκαρυώτες, στην εξαρτώμενη από RNA πολυμεράση II μεταγραφή, υπάρχουν έξι γενικοί μεταγραφικοί παράγοντες: TFIIA, TFIIB (ένα ορθόλογος του αρχαϊκού TFB), TFIID (ένας παράγοντας πολλαπλών υπομονάδων στον οποίο η βασική υπομονάδα, TBP, είναι ένας ορθόλογος της αρχαϊκής TBP), TFIIE (ένας ορθόλογος της αρχαϊκής TFE), TFIIF και TFIIH. Το TFIID είναι το πρώτο συστατικό που συνδέεται με το DNA λόγω δέσμευσης της TBP, ενώ το TFIIH είναι το τελευταίο συστατικό που στρατολογείται. Στα αρχαία και στους ευκαρυώτες, το κλειστό σύμπλεγμα RNA πολυμεράσης-προαγωγέα αναφέρεται συνήθως ως σύμπλεγμα προεκκίνησης.[36] Η έναρξη της μεταγραφής ρυθμίζεται από επιπρόσθετες πρωτεΐνες, γνωστές ως ενεργοποιητές και καταστολείς (repressors), και, σε ορισμένες περιπτώσεις, σχετιζόμενες με συνενεργοποιητές (coactivators) ή συνκαταστολείς (corepressors), οι οποίοι ρυθμίζουν το σχηματισμό και τη λειτουργία του συμπλέγματος έναρξης μεταγραφής.[6]
Διαφυγή προαγωγέων (υποκινητών)
[Επεξεργασία | επεξεργασία κώδικα]Αφού συντεθεί ο πρώτος δεσμός, η RNA πολυμεράση πρέπει να διαφύγει από τον προαγωγέα (υποκινητή). Κατά τη διάρκεια αυτής της περιόδου υπάρχει μια τάση απελευθέρωσης του μεταγραφήματος RNA και παραγωγής περικομμένων μεταγραφημάτων. Αυτό ονομάζεται ανεπιτυχής έναρξη (abortive initiation) και είναι κοινή τόσο για ευκαρυώτες όσο και για προκαρυώτες.[37] Η ανεπιτυχής έναρξη συνεχίζει να συμβαίνει έως ότου συντεθεί ένα προϊόν RNA μήκους κατωφλίου περίπου 10 νουκλεοτιδίων, οπότε λαμβάνει χώρα διαφυγή προαγωγέα και σχηματίζεται ένα σύμπλοκο επιμήκυνσης μεταγραφής. Μηχανιστικά, η διαφυγή του προαγωγέα λαμβάνει χώρα μέσω του τσαλακώματος του DNA (DNA scrunching), παρέχοντας την ενέργεια που απαιτείται για το σπάσιμο των αλληλεπιδράσεων μεταξύ του ολοενζύμου RNA πολυμεράσης και του προαγωγέα.[38] Στα βακτήρια, θεωρούνταν ιστορικά ότι ο παράγοντας σίγμα απελευθερώνεται οπωσδήποτε μετά την κάθαρση του προαγωγέα. Αυτή η θεωρία ήταν γνωστή ως πρότυπο υποχρεωτικής απελευθέρωσης. Ωστόσο, μεταγενέστερα δεδομένα έδειξαν ότι κατά και μετά την εκκαθάριση του προαγωγέα, ο παράγοντας σίγμα απελευθερώνεται σύμφωνα με ένα στοχαστικό πρότυπο γνωστό ως πρότυπο στοχαστικής απελευθέρωσης.[39] Στους ευκαρυώτες, σε έναν εξαρτώμενο από RNA πολυμεράση II προαγωγέα, κατά την κάθαρση του προαγωγέα, ο TFIIH φωσφορυλιώνει τη σερίνη 5 στην καρβοξυτελική περιοχή της RNA πολυμεράσης II, οδηγώντας στη στρατολόγηση του ενζύμου κάλυψης (capping enzyme, CE).[40][41] Ο ακριβής μηχανισμός του τρόπου με τον οποίο το CE επάγει την κάθαρση του προαγωγέα στους ευκαρυώτες δεν είναι ακόμη γνωστός.
Επιμήκυνση
[Επεξεργασία | επεξεργασία κώδικα]
Ένας κλώνος του DNA, ο κλώνος-εκμαγείο (ή μη κωδικοποιητικός κλώνος), χρησιμοποιείται ως πρότυπο για τη σύνθεση RNA. Καθώς προχωρά η μεταγραφή, η πολυμεράση RNA διασχίζει τον κλώνο του εκμαγείου και χρησιμοποιεί τη συμπληρωματικότητα του ζεύγους βάσεων με το πρότυπο DNA για να δημιουργήσει ένα αντίγραφο RNA (το οποίο επιμηκύνεται κατά τη διάρκεια της διέλευσης). Αν και η RNA πολυμεράση διασχίζει τον κλώνο του εκμαγείου από 3' → 5', ο κωδικοποιητικός (μη-πρότυπος) κλώνος και το νεοσχηματισμένο RNA μπορούν επίσης να χρησιμοποιηθούν ως σημεία αναφοράς, επομένως η μεταγραφή μπορεί να περιγραφεί ότι λαμβάνει χώρα κατά την κατεύθυνση 5' → 3'. Αυτό παράγει ένα μόριο RNA από το 5' → 3', ένα ακριβές αντίγραφο του κωδικοποιητικού κλώνου (εκτός από το ότι η θυμίνη αντικαθίσταται με ουρακίλη, και τα νουκλεοτίδια αποτελούνται από ένα σάκχαρο ριβόζης (με 5 άνθρακες), ενώ το DNA έχει δεοξυριβόζη (ένα λιγότερο άτομο οξυγόνου στο φωσφορικό σκελετό του σακχάρου του).[3] Η μεταγραφή του mRNA μπορεί να περιλαμβάνει πολλές RNA πολυμεράσες σε ένα μόνο πρότυπο DNA και πολλούς γύρους μεταγραφής (ενίσχυση συγκεκριμένου mRNA), έτσι πολλά μόρια mRNA μπορούν να παραχθούν γρήγορα από ένα μόνο αντίγραφο ενός γονιδίου. Οι χαρακτηριστικοί ρυθμοί επιμήκυνσης σε προκαρυώτες και ευκαρυώτες είναι περίπου 10-100 nts/s.[42] Στους ευκαρυώτες, ωστόσο, τα νουκλεοσώματα δρουν ως κύριοι φραγμοί στη μεταγραφή πολυμερασών κατά την επιμήκυνση της μεταγραφής.[43][44] Σε αυτούς τους οργανισμούς, η παύση που προκαλείται από τα νουκλεοσώματα μπορεί να ρυθμιστεί από παράγοντες επιμήκυνσης μεταγραφής όπως το TFIIS.[44] Η επιμήκυνση περιλαμβάνει επίσης έναν μηχανισμό διόρθωσης που μπορεί να αντικαταστήσει τις εσφαλμένα ενσωματωμένες βάσεις. Στους ευκαρυώτες, αυτό μπορεί να αντιστοιχεί με μικρές παύσεις κατά τη διάρκεια της μεταγραφής που επιτρέπουν τη δέσμευση κατάλληλων παραγόντων επεξεργασίας RNA. Αυτές οι παύσεις μπορεί να είναι εγγενείς στην RNA πολυμεράση ή να οφείλονται στη δομή της χρωματίνης. Οι θραύσεις διπλού κλώνου σε ενεργά μεταγραμμένες περιοχές του DNA επιδιορθώνονται με ομόλογο ανασυνδυασμό κατά τη διάρκεια των φάσεων S και G2 του κυτταρικού κύκλου.[45][46] Εφόσον η μεταγραφή ενισχύει την προσβασιμότητα του DNA σε εξωγενείς χημικές ουσίες και εσωτερικούς μεταβολίτες που μπορούν να προκαλέσουν ανασυνδυασμογόνες βλάβες, ο ομόλογος ανασυνδυασμός μιας συγκεκριμένης αλληλουχίας DNA μπορεί να διεγείρεται έντονα από τη μεταγραφή.[47]
Τερματισμός
[Επεξεργασία | επεξεργασία κώδικα]Τα βακτήρια χρησιμοποιούν δύο διαφορετικές στρατηγικές για τον τερματισμό της μεταγραφής – τερματισμό ανεξάρτητο από το Rho και τερματισμό εξαρτώμενο από το Rho. Στον ανεξάρτητο από το Rho τερματισμό της μεταγραφής, η μεταγραφή του RNA σταματά όταν το νεοσυντιθέμενο μόριο RNA σχηματίζει έναν πλούσιο σε G-C βρόχο φουρκέτας που ακολουθείται από μια σειρά U. Όταν σχηματίζεται η φουρκέτα, η μηχανική καταπόνηση σπάει τους ασθενείς δεσμούς rU-dA, γεμίζοντας τώρα το υβρίδιο DNA-RNA. Αυτό τραβάει το μεταγράφημα poly-U έξω από την ενεργό θέση της RNA πολυμεράσης, τερματίζοντας τη μεταγραφή. Στoν εξαρτώμενo από Rho τερματισμό, ο παράγοντας Rho, ένας πρωτεϊνικός παράγοντας, αποσταθεροποιεί την αλληλεπίδραση μεταξύ του εκμαγείου και του mRNA, απελευθερώνοντας έτσι το νεοσυντιθέμενο mRNA από το σύμπλεγμα επιμήκυνσης.[48] Ο τερματισμός της μεταγραφής στους ευκαρυώτες είναι λιγότερο κατανοητός από ότι στα βακτήρια, αλλά περιλαμβάνει διάσπαση του νέου μεταγραφήματος που ακολουθείται από ανεξάρτητη από το εκμαγείο προσθήκη αδενινών στο νέο 3' άκρο του, σε μια διαδικασία που ονομάζεται πολυαδενυλίωση.[49] Πέρα από τον τερματισμό από αλληλουχίες τερματισμού (που είναι μέρος ενός γονιδίου), η μεταγραφή μπορεί επίσης να χρειαστεί να τερματιστεί όταν αντιμετωπίζει καταστάσεις όπως βλάβη του DNA ή ενεργή διχάλα αντιγραφής (replication fork). Στα βακτήρια, η ATPάση στο γονίδιο μείωσης της συχνότητας μετάλλαξης (Mutation Frequency Decline, Mfd) μπορεί να αφαιρέσει μια πολυμεράση RNA που έχει σταματήσει σε μια βλάβη ανοίγοντας τον σφιγκτήρα της. Επιστρατεύει επίσης μηχανισμούς επιδιόρθωσης αποκοπής νουκλεοτιδίων για την αποκατάσταση της βλάβης. Το Mfd προτείνεται επίσης για την επίλυση των συγκρούσεων μεταξύ της αντιγραφής του DNA και της μεταγραφής.[50] Στους ευκαυρώτες, η ATPάση TTF2 βοηθά στην καταστολή της δράσης των RNAP I και II κατά τη διάρκεια της μίτωσης, αποτρέποντας σφάλματα στον χρωμοσωμικό διαχωρισμό.[51] Στα αρχαία, η Eta ATPάση προτείνεται ότι διαδραματίζει παρόμοιο ρόλο.[52]
Η μεταγραφή αυξάνει την ευαισθησία σε βλάβη του DNA
[Επεξεργασία | επεξεργασία κώδικα]Η βλάβη του γονιδιώματος εμφανίζεται με υψηλή συχνότητα, η οποία εκτιμάται ότι κυμαίνεται μεταξύ δεκάδων και εκατοντάδων χιλιάδων βλαβών στο DNA που προκύπτουν σε κάθε κύτταρο κάθε μέρα.[53] Η διαδικασία μεταγραφής είναι μια κύρια πηγή βλάβης του DNA, λόγω του σχηματισμού ενδιάμεσων μονόκλωνων DNA που είναι ευάλωτα σε βλάβες.[53] Η ρύθμιση της μεταγραφής με διαδικασίες που χρησιμοποιούν επιδιόρθωση εκτομής βάσεων ή/και τοποϊσομερασών για την αποκοπή και την αναδιαμόρφωση του γονιδιώματος αυξάνει επίσης την τρωτότητα του DNA σε βλάβες.[53]
Ο ρόλος της RNA πολυμεράσης στις μετα-μεταγραφικές αλλαγές στο RNA
[Επεξεργασία | επεξεργασία κώδικα]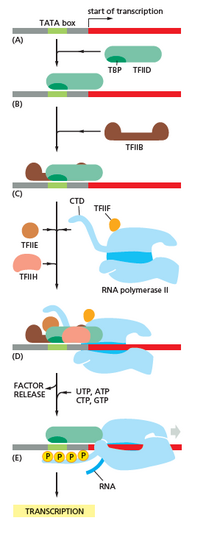
Η RNA πολυμεράση παίζει πολύ κρίσιμο ρόλο σε όλα τα στάδια, συμπεριλαμβανομένων των μετα-μεταγραφικών αλλαγών στο RNA.
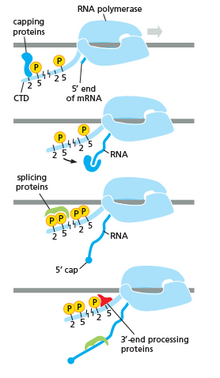
Όπως φαίνεται στην εικόνα στα δεξιά, είναι προφανές ότι το CTD (C Terminal Domain) είναι μια ουρά που αλλάζει το σχήμα του. Αυτή η ουρά θα χρησιμοποιηθεί ως φορέας ματίσματος, κάλυψης και πολυαδενυλίωσης, όπως φαίνεται στην εικόνα στα αριστερά.[54]
Αναστολείς
[Επεξεργασία | επεξεργασία κώδικα]Οι αναστολείς μεταγραφής μπορούν να χρησιμοποιηθούν ως αντιβιοτικά κατά, για παράδειγμα, παθογόνων βακτηρίων (αντιβακτηριακά) και μυκήτων (αντιμυκητιακά). Ένα παράδειγμα τέτοιου αντιβακτηριακού είναι η ριφαμπικίνη, η οποία αναστέλλει τη βακτηριακή μεταγραφή του DNA σε mRNA αναστέλλοντας την εξαρτώμενη από το DNA RNA πολυμεράση δεσμεύοντας τη βήτα-υπομονάδα της, ενώ η 8-υδροξυκινολίνη είναι ένας αντιμυκητιακός αναστολέας μεταγραφής.[55] Τα αποτελέσματα της μεθυλίωσης της ιστόνης μπορεί επίσης να λειτουργήσουν για να αναστέλλουν τη δράση της μεταγραφής. Ισχυρά, βιοδραστικά φυσικά προϊόντα όπως η τριπτολίδη που αναστέλλουν τη μεταγραφή σε θηλαστικά μέσω της αναστολής της υπομονάδας XPB του γενικού μεταγραφικού παράγοντα TFIIH έχει πρόσφατα αναφερθεί ως συζυγές γλυκόζης για τη στόχευση υποξικών καρκινικών κυττάρων με αυξημένη παραγωγή μεταφορέα γλυκόζης.[56]
Ενδογενείς αναστολείς
[Επεξεργασία | επεξεργασία κώδικα]Στα σπονδυλωτά, η πλειοψηφία των γονιδίων προαγωγέων περιέχει μια νησίδα CpG με πολυάριθμες θέσεις CpG.[57] Όταν πολλές από τις θέσεις CpG προαγωγέα ενός γονιδίου μεθυλιώνονται το γονίδιο αναστέλλεται (αποσιωπάται).[58] Οι καρκίνοι του παχέος εντέρου έχουν τυπικά 3 έως 6 οδηγούς μεταλλάξεων και 33 έως 66 μεταλλάξεις ωτοστόπ (hitchhiker) ή επιβατών.[59] Ωστόσο, η μεταγραφική αναστολή (σίγαση) μπορεί να έχει μεγαλύτερη σημασία από τη μετάλλαξη στην πρόκληση εξέλιξης σε καρκίνο. Για παράδειγμα, σε καρκίνους του παχέος εντέρου περίπου 600 έως 800 γονίδια αναστέλλονται μεταγραφικά από τη μεθυλίωση της νησίδας CpG. Η μεταγραφική καταστολή στον καρκίνο μπορεί επίσης να συμβεί από άλλους επιγενετικούς μηχανισμούς, όπως η αλλοιωμένη παραγωγή των μικροRNAs.[60] Στον καρκίνο του μαστού, η μεταγραφική καταστολή του BRCA1 μπορεί να συμβεί πιο συχνά από υπερπαραγωγή μικροRNA-182 παρά από υπερμεθυλίωση του προαγωγέα BRCA1.
Εργοστάσια μεταγραφής
[Επεξεργασία | επεξεργασία κώδικα]Οι ενεργές μονάδες μεταγραφής συγκεντρώνονται στον πυρήνα, σε διακριτές τοποθεσίες που ονομάζονται εργοστάσια μεταγραφής (Transcription factories) ή ευχρωματίνη (euchromatin). Τέτοιες θέσεις μπορούν να οπτικοποιηθούν επιτρέποντας στις δεσμευμένες πολυμεράσες να επεκτείνουν τα μεταγραφήματά τους σε επισημασμένους προδρόμους (Br-UTP ή Br-U) και ανοσοεπισήμανση του επισημασμένου εκκολαπτόμενου RNA. Τα εργοστάσια μεταγραφής μπορούν επίσης να εντοπιστούν χρησιμοποιώντας υβριδισμό φθορισμού in situ ή να επισημανθούν από αντισώματα που κατευθύνονται κατά των πολυμερασών. Υπάρχουν ≈10.000 εργοστάσια στο νουκλεόπλασμα ενός κυττάρου HeLa, μεταξύ των οποίων είναι ≈8.000 εργοστάσια πολυμεράσης II και ≈2.000 εργοστάσια πολυμεράσης III. Κάθε εργοστάσιο πολυμεράσης II περιέχει ≈8 πολυμεράσες. Καθώς οι περισσότερες ενεργές μονάδες μεταγραφής συνδέονται με μία μόνο πολυμεράση, κάθε εργοστάσιο περιέχει συνήθως ≈8 διαφορετικές μεταγραφικές μονάδες. Αυτές οι μονάδες μπορεί να συσχετίζονται μέσω προαγωγέων και/ή ενισχυτών, με βρόχους που σχηματίζουν ένα σύννεφο γύρω από τον παράγοντα.[61]
Ιστορικό
[Επεξεργασία | επεξεργασία κώδικα]Ένα μόριο που επιτρέπει στο γενετικό υλικό να πραγματοποιηθεί ως πρωτεΐνη υποτέθηκε για πρώτη φορά από τους Φρανσουά Ζακόμπ και Ζακ Μονό. Ο Severo Ochoa κέρδισε Βραβείο Νόμπελ Φυσιολογίας και Ιατρικής το 1959 για την ανάπτυξη μιας διαδικασίας σύνθεσης RNA in vitro με πολυνουκλεοτιδική φωσφορυλάση, η οποία ήταν χρήσιμη για τη διάσπαση του γενετικού κώδικα. Η σύνθεση RNA από RNA πολυμεράση καθιερώθηκε "in vitro" από πολλά εργαστήρια μέχρι το 1965. Ωστόσο, το RNA που συντίθεται από αυτά τα ένζυμα είχε ιδιότητες που υποδηλώνουν την ύπαρξη ενός πρόσθετου παράγοντα που απαιτείται για να τερματιστεί σωστά η μεταγραφή. Ο Roger D. Kornberg κέρδισε το 2006 Βραβείο Νόμπελ Χημείας "για τις μελέτες του σχετικά με τη μοριακή βάση της ευκαρυωτικής μεταγραφής".[62]
Μέτρηση και ανίχνευση
[Επεξεργασία | επεξεργασία κώδικα]
Η μεταγραφή μπορεί να μετρηθεί και να ανιχνευθεί με διάφορους τρόπους
- Προσδιορισμός μεταγραφής με κασέτα χωρίς γουανίνη (G-Less Cassette): μετρά την ισχύ του προαγωγέα (υποκινητή)
- Προσδιορισμός μεταγραφής Run-off: προσδιορίζει τοποθεσίες έναρξης μεταγραφής (transcription start sites, TSS)
- Προσδιορισμός Nuclear run-on: μετρά τη σχετική αφθονία των νεοσχηματισμένων μεταγραφημάτων
- KAS-seq: μετρά μονόκλωνο DNA που παράγεται από RNA πολυμεράσες. Μπορεί να λειτουργήσει με 1.000 κύτταρα.[63]
- Δοκιμασία προστασίας ριβονουκλεάσης (RNase protection assay) and ChIP-Chip της RNA πολυμεράσης: εντοπισμός ενεργών θέσεων μεταγραφής
- RT-PCR: μετρά την απόλυτη αφθονία των επιπέδων ολικού ή πυρηνικού RNA, τα οποία ωστόσο μπορεί να διαφέρουν από τους ρυθμούς μεταγραφής
- Μικροσυστοιχίες DNA: μετρούν τη σχετική αφθονία των γενικών επιπέδων ολικού ή πυρηνικού RNA• Ωστόσο, αυτά μπορεί να διαφέρουν από τα ποσοστά μεταγραφής
- Φθορίζων επιτόπιος υβριδισμός (In situ hybridization): ανιχνεύει την παρουσία μεταγραφήματος
- Επισήμανση MS2: με την ενσωμάτωση βρόχων κορμών (stem loops) του RNA, όπως το MS2, σε ένα γονίδιο, αυτά ενσωματώνονται στο νεοσυντιθέμενο RNA. Οι βρόχοι κορμού μπορούν στη συνέχεια να ανιχνευθούν χρησιμοποιώντας μια σύντηξη του GFP και της πρωτεΐνης περιβλήματος MS2, η οποία έχει υψηλή συγγένεια, ειδική για την αλληλουχία αλληλεπίδραση με τους βρόχους στελέχους MS2. Η στρατολόγηση του GFP στη θέση της μεταγραφής οπτικοποιείται ως ένα μόνο φθορίζον σημείο. Αυτή η νέα προσέγγιση έχει αποκαλύψει ότι η μεταγραφή λαμβάνει χώρα σε ασυνεχείς εκρήξεις ή παλμούς. Με την αξιοσημείωτη εξαίρεση των επιτόπιων τεχνικών, οι περισσότερες άλλες μέθοδοι παρέχουν μέσους όρους κυτταρικού πληθυσμού και δεν είναι ικανές να ανιχνεύσουν αυτή τη θεμελιώδη ιδιότητα των γονιδίων.[64]
- Στύπωμα Northern (Northern blot) η παραδοσιακή μέθοδος, και μέχρι την εμφάνιση του RNA-Seq, η πιο ποσοτική
- Αλληλούχιση RNA (RNA-Seq): εφαρμόζει τεχνικές αλληλούχισης επόμενης γενιάς για την αλληλουχία ολόκληρων μεταγραφωμάτων (transscriptomes), που επιτρέπει τη μέτρηση της σχετικής αφθονίας του RNA, καθώς και την ανίχνευση πρόσθετων παραλλαγών όπως γονίδια σύντηξης, μετα-μεταγραφικές επεξεργασίες και νέες τοποθεσίες ματίσματος
- Αλληλούχιση μεμονωμένου κυττάρου (Single-cell RNA-sequencing): ενισχύει και διαβάζει μερικά μεταγραφώματα από απομονωμένα κύτταρα, επιτρέποντας λεπτομερείς αναλύσεις του RNA σε ιστούς, έμβρυα και καρκίνους
Αντίστροφη μεταγραφή
[Επεξεργασία | επεξεργασία κώδικα]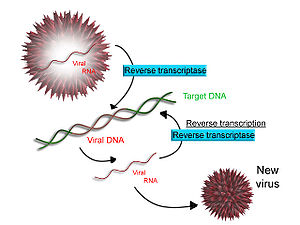
Ορισμένοι ιοί (όπως ο HIV, η αιτία του AIDS), έχουν την ικανότητα να μεταγράφουν το RNA σε DNA. Ο HIV έχει ένα γονιδίωμα RNA που είναι "αντίστροφα μεταγραμμένο" σε DNA. Το DNA που προκύπτει μπορεί να συγχωνευθεί με το γονιδίωμα του DNA του κυττάρου ξενιστή. Το κύριο ένζυμο που είναι υπεύθυνο για τη σύνθεση του DNA από ένα πρότυπο RNA ονομάζεται αντίστροφη μεταγραφάση.[65] Στην περίπτωση του HIV, η αντίστροφη μεταγραφάση είναι υπεύθυνη για τη σύνθεση ενός κλώνου συμπληρωματικού DNA (cDNA) στο γονιδίωμα του ιικού RNA. Το ένζυμο ριβονουκλεάση Η στη συνέχεια χωνεύει τον κλώνο RNA και η αντίστροφη μεταγραφάση συνθέτει έναν συμπληρωματικό κλώνο DNA για να σχηματίσει μια δομή DNA διπλής έλικας (cDNA). Το cDNA ενσωματώνεται στο γονιδίωμα του κυττάρου ξενιστή από το ένζυμο ενσωματάση (integrase), το οποίο αναγκάζει το κύτταρο ξενιστή να παράγει ιικές πρωτεΐνες που επανασυναρμολογούνται σε νέα ιικά σωματίδια. Στον HIV, μετά από αυτό, το κύτταρο ξενιστής υφίσταται προγραμματισμένο κυτταρικό θάνατο, ή απόπτωση, των Τ κυττάρων.[66] Ωστόσο, σε άλλους ρετροϊούς, το κύτταρο ξενιστής παραμένει άθικτο καθώς ο ιός βγαίνει από το κύτταρο. Ορισμένα ευκαρυωτικά κύτταρα περιέχουν ένα ένζυμο με αντίστροφη μεταγραφική δραστηριότητα που ονομάζεται τελομεράση (telomerase). Η τελομεράση φέρει ένα πρότυπο RNA από το οποίο συνθέτει ένα τελομερές, μια επαναλαμβανόμενη αλληλουχία DNA, μέχρι το τέλος των γραμμικών χρωμοσωμάτων. Είναι σημαντικό γιατί κάθε φορά που ένα γραμμικό χρωμόσωμα διπλασιάζεται, μικραίνει. Με το τελομερές στα άκρα των χρωμοσωμάτων, η βράχυνση εξαλείφει μέρος της μη βασικής, επαναλαμβανόμενης αλληλουχίας, αντί της αλληλουχίας DNA που κωδικοποιεί την πρωτεΐνη πιο μακριά από το άκρο του χρωμοσώματος. Η τελομεράση ενεργοποιείται συχνά σε καρκινικά κύτταρα για να επιτρέψει στα καρκινικά κύτταρα να αντιγράψουν τα γονιδιώματά τους επ' αόριστον χωρίς να χάσουν σημαντική αλληλουχία DNA που κωδικοποιεί πρωτεΐνες. Η ενεργοποίηση της τελομεράσης θα μπορούσε να είναι μέρος της διαδικασίας που επιτρέπει στα καρκινικά κύτταρα να γίνουν αθάνατα. Ο παράγοντας αθανασίας του καρκίνου μέσω της επιμήκυνσης των τελομερών λόγω της τελομεράσης έχει αποδειχθεί ότι εμφανίζεται στο 90% όλων των καρκινογόνων όγκων in vivo με το υπόλοιπο 10% να χρησιμοποιεί μια εναλλακτική οδό συντήρησης τελομερών που ονομάζεται ALT ή εναλλακτική επιμήκυνση των τελομερών (Alternative Lengthening of Telomeres, ALT).[67]
Σημειώσεις
[Επεξεργασία | επεξεργασία κώδικα]- ↑ Σημαντικοί ιοί −ssRNA σπονδυλωτών περιλαμβάνουν τον ιό Έμπολα, τους ιούς hanta, τους ιούς της γρίπης, τον ιό του πυρετού Lassa και τον ιό της λύσσας.
Παραπομπές
[Επεξεργασία | επεξεργασία κώδικα]- ↑ «Tentative identification of RNA-dependent RNA polymerases of dsRNA viruses and their relationship to positive strand RNA viral polymerases». FEBS Letters 252 (1–2): 42–6. July 1989. doi:. PMID 2759231. Bibcode: 1989FEBSL.252...42K.
- ↑ 2,0 2,1 Eldra P. Solomon, Linda R. Berg, Diana W. Martin. Biology, 8th Edition, International Student Edition. Thomson Brooks/Cole. (ISBN 978-0495317142)
- ↑ 3,0 3,1 Clark, David P. (24 Ιουνίου 2005). Molecular Biology (στα Αγγλικά). Elsevier. σελ. 134. ISBN 978-0-08-045421-4.
- ↑ «DNA Strands». www.sci.sdsu.edu. Αρχειοθετήθηκε από το πρωτότυπο στις 27 Οκτωβρίου 2017. Ανακτήθηκε στις 1 Μαΐου 2018.
- ↑ Berg J, Tymoczko JL, Stryer L (2006). Biochemistry (6th έκδοση). San Francisco: W. H. Freeman. ISBN 0-7167-8724-5.
- ↑ 6,0 6,1 6,2 6,3 6,4 6,5 6,6 Watson JD, Baker TA, Bell SP, Gann AA, Levine M, Losick RM (2013). Molecular Biology of the Gene (7th έκδοση). Pearson. ISBN 978-0-321-76243-6. OCLC 0321762436.
- ↑ «Eukaryotic core promoters and the functional basis of transcription initiation». Nat Rev Mol Cell Biol 19 (10): 621–637. October 2018. doi:. PMID 29946135.
- ↑ «The Why of YY1: Mechanisms of Transcriptional Regulation by Yin Yang 1». Front Cell Dev Biol 8: 592164. 2020. doi:. PMID 33102493.
- ↑ «Transcription factors: from enhancer binding to developmental control». Nat Rev Genet 13 (9): 613–26. September 2012. doi:. PMID 22868264.
- ↑ 10,0 10,1 «Three-dimensional genome restructuring across timescales of activity-induced neuronal gene expression». Nat Neurosci 23 (6): 707–717. June 2020. doi:. PMID 32451484.
- ↑ 11,0 11,1 «Long-range enhancer-promoter contacts in gene expression control». Nat Rev Genet 20 (8): 437–455. August 2019. doi:. PMID 31086298.
- ↑ «Enhancers: five essential questions». Nat Rev Genet 14 (4): 288–95. April 2013. doi:. PMID 23503198.
- ↑ «YY1 Is a Structural Regulator of Enhancer-Promoter Loops». Cell 171 (7): 1573–88.e28. December 2017. doi:. PMID 29224777.
- ↑ «The Human Transcription Factors». Cell 172 (4): 650–665. February 2018. doi:. PMID 29425488.
- ↑ 15,0 15,1 «Positional specificity of different transcription factor classes within enhancers». Proc Natl Acad Sci U S A 115 (30): E7222–30. July 2018. doi:. PMID 29987030. Bibcode: 2018PNAS..115E7222G.
- ↑ «The Mediator complex: a central integrator of transcription». Nat Rev Mol Cell Biol 16 (3): 155–66. March 2015. doi:. PMID 25693131.
- ↑ «The degree of enhancer or promoter activity is reflected by the levels and directionality of eRNA transcription». Genes Dev 32 (1): 42–57. January 2018. doi:. PMID 29378788.
- ↑ «MAP kinase phosphorylation-dependent activation of Elk-1 leads to activation of the co-activator p300». EMBO J 22 (2): 281–91. January 2003. doi:. PMID 12514134.
- ↑ «Enhancer RNAs predict enhancer-gene regulatory links and are critical for enhancer function in neuronal systems». Nucleic Acids Res 48 (17): 9550–70. September 2020. doi:. PMID 32810208.
- ↑ «DNA methylation in human epigenomes depends on local topology of CpG sites». Nucleic Acids Res 44 (11): 5123–32. June 2016. doi:. PMID 26932361.
- ↑ «Cytosine methylation and CpG, TpG (CpA) and TpA frequencies». Gene 333: 143–9. May 2004. doi:. PMID 15177689.
- ↑ «Pervasive and CpG-dependent promoter-like characteristics of transcribed enhancers». Nucleic Acids Res 48 (10): 5306–17. June 2020. doi:. PMID 32338759.
- ↑ «DNA methylation patterns and epigenetic memory». Genes Dev 16 (1): 6–21. January 2002. doi:. PMID 11782440.
- ↑ 24,0 24,1 24,2 «Methyl-CpG-binding domain proteins: readers of the epigenome». Epigenomics 7 (6): 1051–73. 2015. doi:. PMID 25927341.
- ↑ «A census of human transcription factors: function, expression and evolution». Nat. Rev. Genet. 10 (4): 252–63. April 2009. doi:. PMID 19274049.
- ↑ 26,0 26,1 26,2 26,3 «EGR1 recruits TET1 to shape the brain methylome during development and upon neuronal activity». Nat Commun 10 (1): 3892. August 2019. doi:. PMID 31467272. Bibcode: 2019NatCo..10.3892S.
- ↑ 27,0 27,1 «Genome-wide investigation of in vivo EGR-1 binding sites in monocytic differentiation». Genome Biol 10 (4): R41. 2009. doi:. PMID 19374776.
- ↑ «Neuronal DNA Methyltransferases: Epigenetic Mediators between Synaptic Activity and Gene Expression?». Neuroscientist 24 (2): 171–185. April 2018. doi:. PMID 28513272.
- ↑ «Rescue of aging-associated decline in Dnmt3a2 expression restores cognitive abilities». Nat Neurosci 15 (8): 1111–3. July 2012. doi:. PMID 22751036.
- ↑ «The Dnmt3a PWWP domain reads histone 3 lysine 36 trimethylation and guides DNA methylation». J Biol Chem 285 (34): 26114–20. August 2010. doi:. PMID 20547484.
- ↑ «Isoform-specific localization of DNMT3A regulates DNA methylation fidelity at bivalent CpG islands». EMBO J 36 (23): 3421–34. December 2017. doi:. PMID 29074627.
- ↑ «H3K36me2/3 Binding and DNA Binding of the DNA Methyltransferase DNMT3A PWWP Domain Both Contribute to its Chromatin Interaction». J Mol Biol 431 (24): 5063–74. December 2019. doi:. PMID 31634469.
- ↑ «Synaptic control of DNA methylation involves activity-dependent degradation of DNMT3A1 in the nucleus». Neuropsychopharmacology 45 (12): 2120–30. November 2020. doi:. PMID 32726795.
- ↑ 34,0 34,1 Pakay, Julian· Duivenvoorden, Hendrika· Shafee, Thomas· Clarke, Kaitlin (2023). Threshold Concepts in Biochemistry. La Trobe eBureau. doi:10.26826/1017. ISBN 978-0-6484681-9-6. Unknown parameter
|s2cid=ignored (βοήθεια) - ↑ Boutard, Magali (2016). «Global repositioning of transcription start sites in a plant-fermenting bacterium». Nature Communications 7: 13783. doi:. PMID 27982035. Bibcode: 2016NatCo...713783B.
- ↑ Roeder, Robert G. (1991). «The complexities of eukaryotic transcription initiation: regulation of preinitiation complex assembly». Trends in Biochemical Sciences 16 (11): 402–8. doi:. PMID 1776168.
- ↑ «Direct detection of abortive RNA transcripts in vivo». Science 324 (5929): 927–8. May 2009. doi:. PMID 19443781. Bibcode: 2009Sci...324..927G.
- ↑ «Abortive initiation and productive initiation by RNA polymerase involve DNA scrunching». Science 314 (5802): 1139–43. November 2006. doi:. PMID 17110577. Bibcode: 2006Sci...314.1139R.
- ↑ «Holoenzyme switching and stochastic release of sigma factors from RNA polymerase in vivo». Molecular Cell 20 (3): 357–66. November 2005. doi:. PMID 16285918.
- ↑ «Functional interactions of RNA-capping enzyme with factors that positively and negatively regulate promoter escape by RNA polymerase II». Proceedings of the National Academy of Sciences of the United States of America 101 (20): 7572–7. May 2004. doi:. PMID 15136722. Bibcode: 2004PNAS..101.7572M.
- ↑ «Transcription factors IIE and IIH and ATP hydrolysis direct promoter clearance by RNA polymerase II». Cell 77 (1): 145–56. April 1994. doi:. PMID 8156590.
- ↑ Milo, Ron· Philips, Rob (2015). «4. Rates and Duration: Central dogma: Which is faster:transcription or translation?». [[[:Πρότυπο:GBurl]] Cell Biology by the Numbers] Check
|url=value (βοήθεια). CRC Press. σελίδες 231–6. ISBN 978-1-317-23069-4. OCLC 1105558425. - ↑ «Nucleosomal fluctuations govern the transcription dynamics of RNA polymerase II». Science 325 (5940): 626–8. July 2009. doi:. PMID 19644123. Bibcode: 2009Sci...325..626H.
- ↑ 44,0 44,1 «Nucleosomal arrangement affects single-molecule transcription dynamics». Proceedings of the National Academy of Sciences 113 (45): 12733–12738. 2016. doi:. PMID 27791062. Bibcode: 2016PNAS..11312733F.
- ↑ «Transcriptionally active chromatin recruits homologous recombination at DNA double-strand breaks». Nat Struct Mol Biol 21 (4): 366–74. April 2014. doi:. PMID 24658350.
- ↑ «RNA transcripts stimulate homologous recombination by forming DR-loops». Nature 594 (7862): 283–8. June 2021. doi:. PMID 33981036. Bibcode: 2021Natur.594..283O.
- ↑ «Recombinogenic effects of DNA-damaging agents are synergistically increased by transcription in Saccharomyces cerevisiae. New insights into transcription-associated recombination». Genetics 165 (2): 457–66. October 2003. doi:. PMID 14573461.
- ↑ «Rho-dependent termination and ATPases in transcript termination». Biochimica et Biophysica Acta (BBA) - Gene Structure and Expression 1577 (2): 251–260. September 2002. doi:. PMID 12213656.
- ↑ «Overlapping pathways dictate termination of RNA polymerase II transcription». Biochimie 89 (10): 1177–82. October 2007. doi:. PMID 17629387.
- ↑ Shi, J; Wen, A; Zhao, M; Jin, S; You, L; Shi, Y; Dong, S; Hua, X και άλλοι. (18 November 2020). «Structural basis of Mfd-dependent transcription termination.». Nucleic Acids Research 48 (20): 11762–11772. doi:. PMID 33068413.
- ↑ Jiang, Y; Liu, M; Spencer, CA; Price, DH (7 May 2004). «Involvement of transcription termination factor 2 in mitotic repression of transcription elongation.». Molecular Cell 14 (3): 375–85. doi:. PMID 15125840.
- ↑ Marshall, CJ; Qayyum, MZ; Walker, JE; Murakami, KS; Santangelo, TJ (9 August 2022). «The structure and activities of the archaeal transcription termination factor Eta detail vulnerabilities of the transcription elongation complex.». Proceedings of the National Academy of Sciences of the United States of America 119 (32): e2207581119. doi:. PMID 35917344. Bibcode: 2022PNAS..11907581M.
- ↑ 53,0 53,1 53,2 «DNA damage and transcription stress». Mol Cell 84 (1): 70–79. January 2024. doi:. PMID 38103560.
- ↑ Cramer, P.; Armache, K.-J.; Baumli, S.; Benkert, S.; Brueckner, F.; Buchen, C.; Damsma, G.E.; Dengl, S. και άλλοι. (June 2008). «Structure of Eukaryotic RNA Polymerases». Annual Review of Biophysics 37 (1): 337–352. doi:. PMID 18573085. http://dx.doi.org/10.1146/annurev.biophys.37.032807.130008.
- ↑ http://www.sigmaaldrich.com/US/en/product/sial/h6878 8-Hydroxyquinoline from SIGMA-ALDRICH. Retrieved 2022-02-15
- ↑ «A Glucose-Triptolide Conjugate Selectively Targets Cancer Cells under Hypoxia». iScience 23 (9): 101536. 2020. doi:. PMID 33083765. Bibcode: 2020iSci...23j1536D.
- ↑ «A genome-wide analysis of CpG dinucleotides in the human genome distinguishes two distinct classes of promoters». Proceedings of the National Academy of Sciences of the United States of America 103 (5): 1412–7. January 2006. doi:. PMID 16432200. Bibcode: 2006PNAS..103.1412S.
- ↑ «DNA methylation patterns and epigenetic memory». Genes & Development 16 (1): 6–21. January 2002. doi:. PMID 11782440.
- ↑ «Cancer genome landscapes». Science 339 (6127): 1546–58. March 2013. doi:. PMID 23539594. Bibcode: 2013Sci...339.1546V.
- ↑ «MicroRNAs in the DNA Damage/Repair Network and Cancer». International Journal of Genomics 2014: 820248. 2014. doi:. PMID 24616890.
- ↑ «TNFα signals through specialized factories where responsive coding and miRNA genes are transcribed». The EMBO Journal 31 (23): 4404–14. November 2012. doi:. PMID 23103767.
- ↑ «Chemistry 2006». Nobel Foundation. Αρχειοθετήθηκε από το πρωτότυπο στις 15 Μαρτίου 2007. Ανακτήθηκε στις 29 Μαρτίου 2007.
- ↑ «Kethoxal-assisted single-stranded DNA sequencing captures global transcription dynamics and enhancer activity in situ». Nature Methods 17 (5): 515–523. April 2020. doi:. PMID 32251394.
- ↑ «Nature, nurture, or chance: stochastic gene expression and its consequences». Cell 135 (2): 216–26. October 2008. doi:. PMID 18957198.
- ↑ Clark, David P. (24 Ιουνίου 2005). Molecular Biology (στα Αγγλικά). Elsevier. σελ. 63. ISBN 978-0-08-045421-4.
- ↑ Kolesnikova IN (2000). «Some patterns of apoptosis mechanism during HIV-infection». Dissertation (στα Ρωσικά). Αρχειοθετήθηκε από το πρωτότυπο στις 10 Ιουλίου 2011. Ανακτήθηκε στις 20 Φεβρουαρίου 2011.
- ↑ «Alternative lengthening of telomeres: models, mechanisms and implications». Nature Reviews Genetics 11 (5): 319–30. May 2010. doi:. PMID 20351727.
Εξωτερικοί σύνδεσμοι
[Επεξεργασία | επεξεργασία κώδικα]- Interactive Java simulation of transcription initiation. Αρχειοθετήθηκε 2011-07-22 στο Wayback Machine. From Center for Models of Life Αρχειοθετήθηκε 2011-08-09 στο Wayback Machine. at the Niels Bohr Institute.
- Interactive Java simulation of transcription interference—a game of promoter dominance in bacterial virus. Αρχειοθετήθηκε 2011-08-26 στο Wayback Machine. From Center for Models of Life Αρχειοθετήθηκε 2011-08-09 στο Wayback Machine. at the Niels Bohr Institute.
- Virtual Cell Animation Collection, Introducing Transcription Αρχειοθετήθηκε 2021-04-14 στο Wayback Machine.